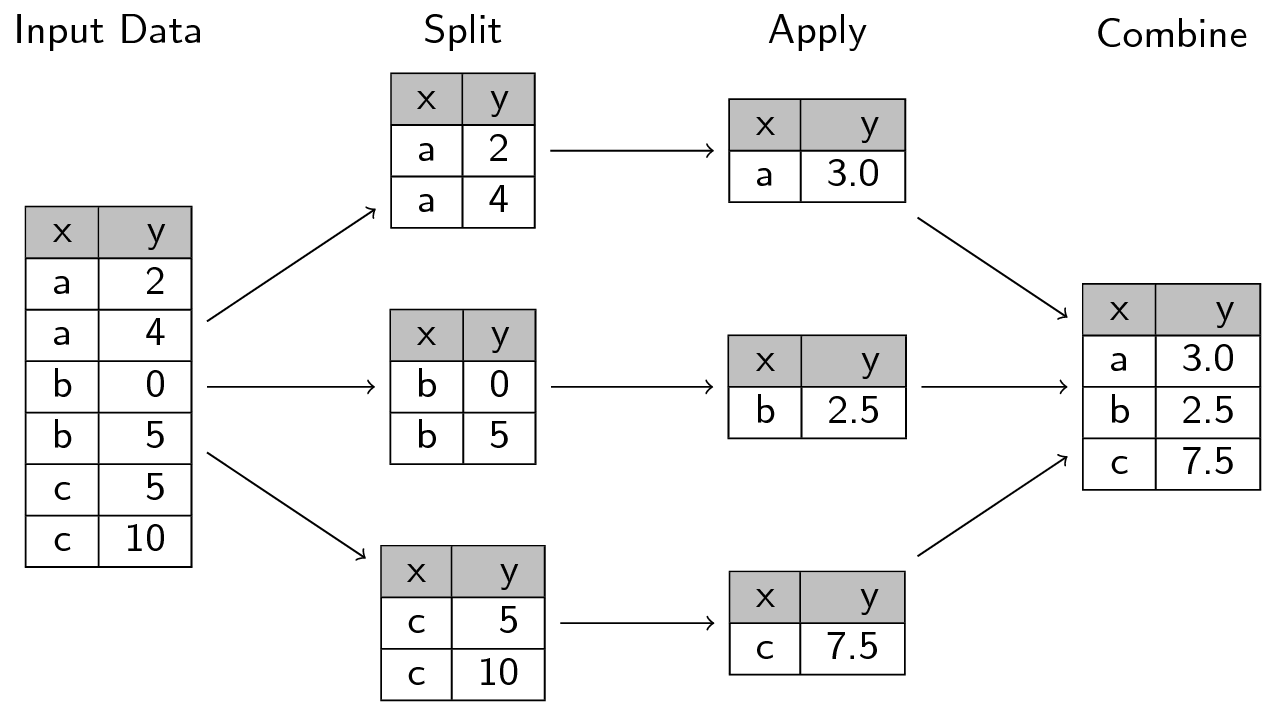
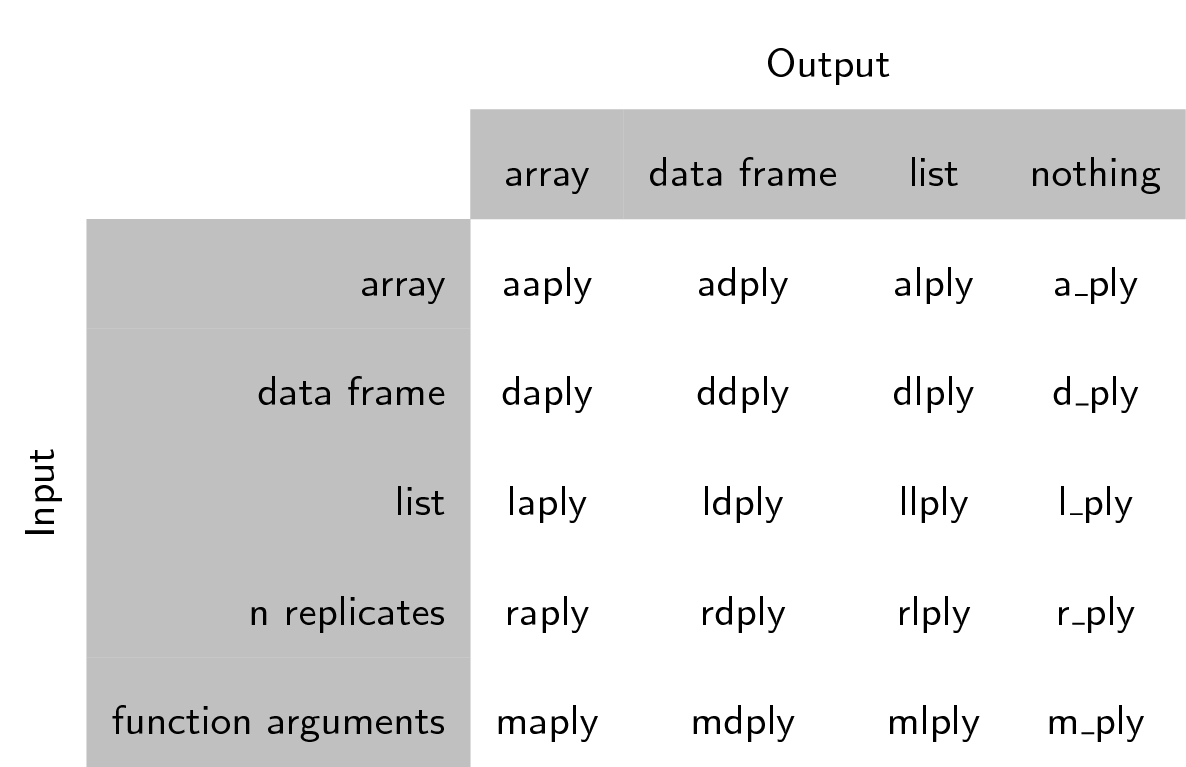
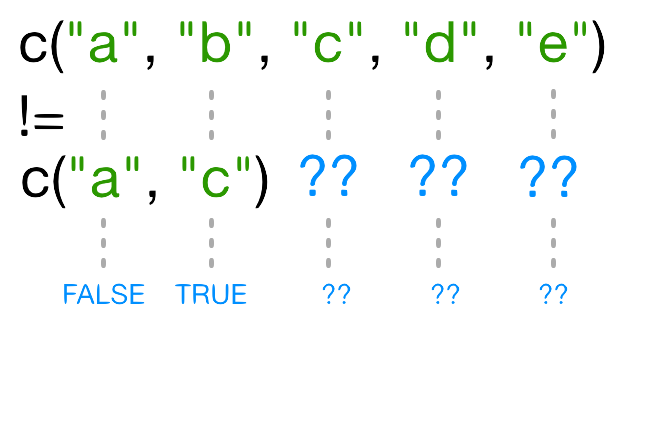| title | author | output | ||||
|---|---|---|---|---|---|---|
R para Analisis Cientificos |
Cristian Farias |
|
Instala los siguientes paquetes: ggplot2, plyr, gapminder, dplyr, tidyr.
Puedes utilizar el comando install.packages() para instalar los paquetes requeridos si no los tienes ya instalados.
if (!requireNamespace("ggplot2", quietly = TRUE)) install.packages("ggplot2")
if (!requireNamespace("plyr", quietly = TRUE)) install.packages("plyr")
if (!requireNamespace("gapminder", quietly = TRUE)) install.packages("gapminder")
if (!requireNamespace("dplyr", quietly = TRUE)) install.packages("dplyr")
if (!requireNamespace("tidyr", quietly = TRUE)) install.packages("tidyr")
library(ggplot2)
library(plyr)
library(gapminder)
library(dplyr)
library(tidyr)-
¿Cómo orientarse en RStudio?
-
¿Cómo interactuar con R?
-
¿Cómo administrar tu entorno?
-
¿Cómo instalar paquetes?
-
Describir el propósito y el uso de cada panel del RStudio IDE
-
Ubicar botones y opciones en RStudio IDE
-
Definir una variable
-
Asignar un dato a una variable
-
Administrar un espacio de trabajo en una sesión R interactiva
-
Usar operadores matemáticos y de comparación
-
Llamar funciones
-
Gestionar paquetes
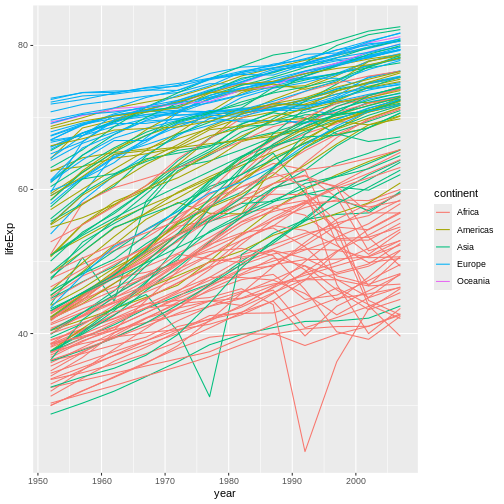
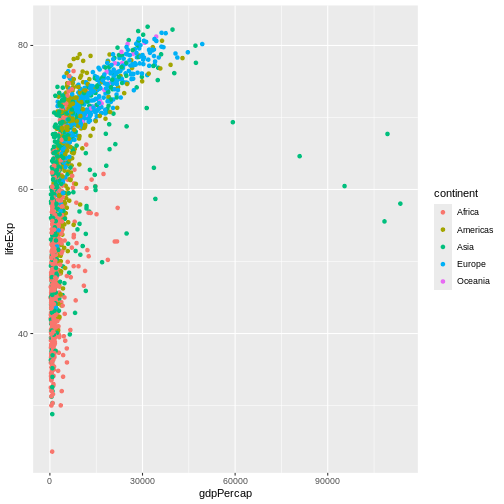
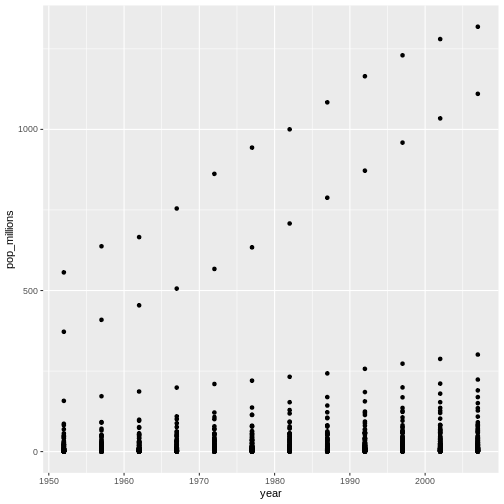
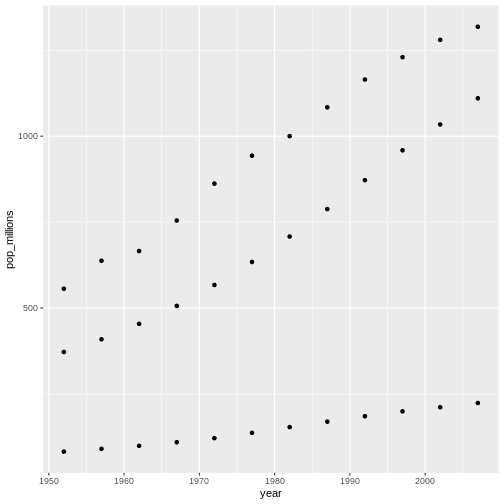
La ciencia es un proceso de varios pasos: una vez que hayas diseñado un experimento y recopilado datos, ¡comienza la verdadera diversión! Esta lección te enseñará cómo comenzar este proceso usando R y RStudio. Comenzaremos con datos brutos, realizaremos análisis exploratorios y aprenderemos a trazar gráficamente los resultados. Este ejemplo comienza con un conjunto de datos de gapminder.org que contiene información sobre la población de muchos países a lo largo del tiempo. ¿Puedes leer estos datos en R? ¿Puedes hacer un gráfico de la población de Senegal? ¿Puedes calcular el ingreso promedio de los países del continente asiático? Al final de estas lecciones, ¡podrás hacer cosas como graficar datos de poblaciones de estos países en menos de un minuto!
Asegúrate de tener instalada la última versión de R y RStudio en tu máquina. Esto es importante, ya que algunos paquetes utilizados en el taller pueden no instalarse correctamente (o no funcionar) si R no está actualizado.
Descarga e instala la última versión de R aquí, descarga e instala RStudio aquí
Bienvenido a la parte R del taller de Software Carpentry.
A lo largo de esta lección, vamos a enseñarte algunos de los fundamentos del lenguaje R, así como algunas buenas prácticas para organizar el código de proyectos científicos que harán tu vida más fácil.
Usaremos RStudio: un entorno de desarrollo integrado y gratuito de código abierto. RStudio proporciona un editor incorporado, funciona en todas las plataformas (incluso en servidores) y ofrece muchas ventajas, como la integración de control de versiones y gestión de proyectos.
Diseño básico
Cuando abres por primera vez el RStudio, serás recibido por tres paneles:
-
La consola interactiva de R (a la izquierda)
-
Ambiente/Historial (en la esquina superior derecha)
-
Archivos/Gráficos/Paquetes/Ayuda/Visor (abajo a la derecha)
Si abres archivos, como los scripts R, también se abrirá un panel de editor en la esquina superior izquierda.
Hay dos formas principales en que uno puede trabajar dentro de RStudio.
- Probar y jugar dentro de la consola interactiva de R y luego copiar el código en un archivo .R para ejecutarlo más tarde.
-
Esto funciona bien cuando se hacen pequeñas pruebas y/o se está comenzando.
-
Rápidamente se vuelve laborioso.
- Comienza a escribir un archivo en .R y usa las teclas de acceso directo de RStudio para ejecutar la línea actual, las líneas seleccionadas o modificadas en la consola interactiva.
-
Esta es una buena forma de comenzar; todo tu código estará guardado para después.
-
Podrás ejecutar el archivo que quieres desde RStudio o mediante la función
source ()de R.
RStudio ofrece gran flexibilidad para ejecutar código desde dentro de la ventana del editor Hay botones, opciones de menú y atajos de teclado. Para ejecutar la línea actual, puedes
- hacer clic en el botón
Runarriba en el panel del Editor, o 2. Seleccionar “Run Lines” desde el menú “Code”, o 3. Presionar Ctrl-Enter en Windows o Linux o Command-Enter en OS X. (Este atajo también se puede hacer colocando el mouse sobre el botón). Para ejecutar un bloque de código, selecciónalo y luego pulsaRun. Si has modificado una línea de código dentro de un bloque que acabas de ejecutar, no es necesario re-seleccionar el bloque yRun, puedes usar el botónRe-run the previous region. Esto ejecutará el bloque de código anterior, incluidas las modificaciones que hayas realizado.
Gran parte de tu tiempo en R lo gastarás en la consola interactiva de R. Aquí es donde ejecutarás todo tu código, y puede ser un entorno útil para probar ideas antes de guardarlas en un script R. La consola en RStudio es la misma que obtendrías si escribieras R en la terminal de shell/linea de comandos.
Lo primero que verás en la sesión interactiva de R es un montón de información, seguido por un “>” y un cursor parpadeante. Esto es similar al entorno de la terminal de shell que aprendiste durante las lecciones de shell: R opera con la misma idea de “leer, evaluar, mostrar” (tú escribes comandos, R intenta ejecutarlos y luego devuelve un resultado).
Lo más simple que podrías hacer con R es aritmética:
1 + 100[1] 101
R te mostrará la respuesta, precedido de un “[1]”. No te preocupes por esto por ahora, lo explicaremos más adelante. Por ahora piensa en eso como parte de la salida.
Al igual que bash, si escribes un comando incompleto R esperará a que lo completes:
> 1 ++
Cada vez que presionas Enter y R te muestra un “+” en lugar de “>”, significa que está esperando que completes el comando. Si deseas cancelar un comando, simplemente presiona “Esc” y RStudio te devolverá el “>” prompt.
Si usas R desde la línea de comandos en lugar de estar dentro de RStudio, debes usar Ctrl + C en lugar deEsc para cancelar el comando. ¡Esto se aplica también a los usuarios de Mac!
La cancelación de un comando no sólo es útil para matar comandos incompletos: también puedes usarlo para decirle a R que deje de ejecutar el código (por ejemplo, si tarda mucho más de lo que esperabas), o para deshacerte del código que estás escribiendo actualmente.
Cuando usas R como calculadora, el orden de las operaciones es el mismo que has aprendido en la escuela.
De mayor a menor precedencia:
-
Paréntesis:
(,) -
Exponente:
^o** -
División:
/ -
Multiplicación:
* -
Suma:
+ -
Resta:
-
3 + 5 * 2[1] 13
Usa paréntesis para agrupar las operaciones a fin de forzar el orden de la evaluación o para aclarar lo que deseas hacer.
(3 + 5) * 2[1] 16
Esto puede ser difícil de manejar cuando no es necesario, pero aclara tus intenciones. Recuerda que otros pueden leer tu código.
(3 + (5 * (2 ^ 2))) # difícil de leer
3 + 5 * 2 ^ 2 # claro, si recuerdas las reglas
3 + 5 * (2 ^ 2) # si olvidas algunas reglas, esto podría ayudarEl texto después de cada línea de código se llama “comentario”. Todo lo que sigue después del símbolo hash (o numeral) # es ignorado por R cuando se ejecuta el código.
Los números pequeños o grandes tienen una notación científica:
2/10000[1] 2e-04
Es la abreviatura de “multiplicado por 10 ^ XX”. Entonces 2e-4 es la abreviatura de 2 * 10^(-4).
Tú también puedes escribir números en notación científica:
5e3 # nota la falta del signo menos aquí[1] 5000
R tiene muchas funciones matemáticas integradas. Para llamar a una función, simplemente escribimos su nombre seguido de paréntesis ( ). Todo lo que escribas dentro de los paréntesis se llaman argumentos de la función:
sin(1) # función trigonométrica[1] 0.841471
log(1) # logaritmo natural[1] 0
log10(10) # logaritmo en base-10[1] 1
exp(0.5) # e^(1/2)[1] 1.648721
No te preocupes si no recuerdas todas las funciones en R. Simplemente puedes buscarlas en Google, o si puedes recordar el comienzo del nombre de la función, puedes usar el tabulador para completar su nombre en RStudio.
Esta es una de las ventajas que RStudio tiene sobre R, tiene capacidades de autocompletado que te permiten buscar funciones, sus argumentos y los valores que toman más fácilmente.
Escribir un ? antes del nombre de un comando abrirá la página de ayuda para ese comando. Además de proporcionar una descripción detallada del comando y cómo funciona, al desplazarse hacia la parte inferior de la página de ayuda generalmente se mostrarán ejemplos que ilustran el uso del comando. Veremos un ejemplo más adelante. Puedes consultar también las guías rápidas disponibles en el sitio de RStudio.
Podemos realizar comparaciones en R:
1 == 1 # igualdad (observa dos signos iguales, se lee como "es igual a")[1] TRUE
1 != 2 # desigualdad (leída como "no es igual a")[1] TRUE
1 < 2 # menor que[1] TRUE
1 <= 1 # menor o igual que[1] TRUE
1 > 0 # mayor que[1] TRUE
1 >= -9 # mayor o igual que[1] TRUE
Una advertencia sobre la comparación de números: nunca debes usar == para comparar dos números a menos que sean enteros (integer es un tipo de datos que específica números enteros).
Las computadoras sólo pueden representar números decimales con un cierto grado de precisión, así que dos números que parecen iguales cuando se muestran por R, pueden tener diferentes representaciones subyacentes y por lo tanto ser diferentes por un pequeño margen de error (llamado tolerancia numérica de la máquina).
En su lugar, debes usar la función all.equal.
Lectura adicional: http://floating-point-gui.de/
Podemos almacenar valores en variables usando el operador de asignación <-. Veamos un ejemplo:
x <- 1/40Observa que la asignación no muestra el valor. En cambio, lo almacena para más adelante en algo llamado variable. x ahora contiene el valor 0.025:
x[1] 0.025
Más precisamente, el valor almacenado es una aproximación decimal de esta fracción, llamado número de coma flotante o floating point.
Busca la pestaña Environment en uno de los paneles de RStudio, y verás que x y su valor han aparecido. Nuestra variable x se puede usar en lugar de un número en cualquier cálculo que espere un número:
log(x)[1] -3.688879
Ten en cuenta que las variables pueden reasignarse, es decir, puedes cambiar el valor almacenado en la variable:
x <- 100x tenía el valor 0.025 y ahora tiene el valor 100.
También, los valores de asignación pueden contener la variable asignada:
x <- x + 1 # observa cómo RStudio actualiza la descripción de x en la pestaña superior derecha
y <- x * 2El lado derecho de la asignación puede ser cualquier expresión de R válida. La expresión del lado derecho se evalúa por completo antes de que se realice la asignación.
Los nombres de las variables pueden contener letras, números, guiones bajos y puntos. No pueden comenzar con un número ni contener espacios en absoluto. Diferentes personas usan diferentes convenciones para nombres largos de variables, estos incluyen
-
puntos.entre.palabras
-
guiones_bajos_entre_palabras
-
MayúsculasMinúsculasParaSepararPalabras
Lo que uses depende de ti, pero sé consistente.
También es posible utilizar el operador = para la asignación:
x = 1/40Esta forma es menos común entre los usuarios R. Lo más importante es ser consistente con el operador que usas. Ocasionalmente hay lugares donde es menos confuso usar <- que =, y es el símbolo más común usado en la comunidad. Entonces la recomendación es usar <-.
De los siguientes ejemplos, ¿Cuáles son nombres de variables válidas en R?
min_height
max.height
_age
.mass
MaxLength
min-length
2widths
celsius2kelvinLos siguientes nombres de variables son válidos en R:
min_height
max.height
MaxLength
celsius2kelvinEl punto al inicio crea una variable oculta:
.massLos siguientes no son nombres de variables válidos en R:
_age
min-length
2widthsEs muy importante tener en cuenta que R es vectorizado, lo que significa que las variables y funciones pueden tener vectores como valores y R puede operar en vectores completos a la vez. En contraste con los conceptos de vectores de física y matemáticas, un vector en R describe un conjunto de valores del mismo tipo de datos en un cierto orden. Por ejemplo:
1:5[1] 1 2 3 4 5
2^(1:5)[1] 2 4 8 16 32
x <- 1:5
2^x[1] 2 4 8 16 32
Esto es increíblemente poderoso; discutiremos esto en una próxima lección.
Hay algunos comandos útiles que puedes usar para interactuar con la sesión de R.
ls listará todas las variables y funciones almacenadas en el entorno global (tu sesión de trabajo en R):
ls()[1] "x" "y"
Al igual que en el shell, ls oculta por defecto cualquier variable o función que comience con un “.”. Para listar todos los objetos, escribe ls(all.names = TRUE)
Ten en cuenta que no se dió ningún argumento a ls, pero sí se necesita poner los paréntesis para decirle a R que llame a la función.
Si escribimos ls nada más, ¡R mostrará el código fuente de esa función!
lsfunction (name, pos = -1L, envir = as.environment(pos), all.names = FALSE,
pattern, sorted = TRUE)
{
if (!missing(name)) {
pos <- tryCatch(name, error = function(e) e)
if (inherits(pos, "error")) {
name <- substitute(name)
if (!is.character(name))
name <- deparse(name)
warning(gettextf("%s converted to character string",
sQuote(name)), domain = NA)
pos <- name
}
}
all.names <- .Internal(ls(envir, all.names, sorted))
if (!missing(pattern)) {
if ((ll <- length(grep("[", pattern, fixed = TRUE))) &&
ll != length(grep("]", pattern, fixed = TRUE))) {
if (pattern == "[") {
pattern <- "\\["
warning("replaced regular expression pattern '[' by '\\\\['")
}
else if (length(grep("[^\\\\]\\[<-", pattern))) {
pattern <- sub("\\[<-", "\\\\\\[<-", pattern)
warning("replaced '[<-' by '\\\\[<-' in regular expression pattern")
}
}
grep(pattern, all.names, value = TRUE)
}
else all.names
}
<bytecode: 0x557005704d80>
<environment: namespace:base>
Puedes usar rm para eliminar objetos que ya no necesitas:
rm(x)Si tienes muchas cosas en tu entorno y deseas borrarlas todas, puedes pasar los resultados de ls y mandarlos a la función rm:
rm(list = ls())En este caso, hemos combinado los dos. Al igual que el orden de las operaciones, todo lo que se encuentre dentro de los paréntesis más internos se evalúa primero, y así sucesivamente.
En este caso, hemos especificado que los resultados de ls se deben usar para el argumento list y luego remover la lista con rm. Cuando asignes valores a argumentos por su nombre, debes usar el operador =.
Si, en cambio, usamos <-, habrá efectos secundarios no deseados, o puedes recibir un mensaje de error:
rm(list <- ls())Error in rm(list <- ls()): ... must contain names or character strings
¡Presta atención cuando R hace algo inesperado! Los errores, como el anterior, se lanzan cuando R no puede proceder a un cálculo. Por otro lado, las advertencias generalmente significan que la función se ha ejecutado, pero probablemente no funcionó como se esperaba.
En ambos casos, el mensaje que muestra R usualmente te da pistas sobre cómo solucionar el problema.
Es posible agregar funciones a R escribiendo un paquete u obteniendo un paquete escrito por otra persona. Hay más de 10.000 paquetes disponibles en CRAN (la red completa de archivos R). R y RStudio tienen funcionalidad para administrar paquetes:
-
Puedes ver qué paquetes están instalados escribiendo
installed.packages() -
Puedes instalar paquetes escribiendo
install.packages("nombre_de_paquete") -
Puedes actualizar los paquetes instalados escribiendo
update.packages() -
Puedes eliminar un paquete con
remove.packages("nombre_de_paquete") -
Puedes hacer que un paquete esté disponible para su uso con
library(nombre_de_paquete)
¿Cuál será el valor de cada variable después de cada comando en el siguiente programa?
mass <- 47.5
age <- 122
mass <- mass * 2.3
age <- age - 20mass <- 47.5Esto dará un valor de 47.5 para la variable mass
age <- 122Esto dará un valor de 122 para la variable age
mass <- mass * 2.3Multiplica el valor existente en mass 47.5 por 2.3 para dar un nuevo valor 109.25 a la variable mass.
age <- age - 20Resta 20 del valor existente de 122 para obtener un nuevo valor de 102 para la variable age.
Ejecuta el código del desafío anterior y escribe un comando para comparar la variable mass con age. ¿Es la variable mass más grande que age?
Una forma de responder esta pregunta en R es usar > para hacer lo siguiente:
mass > age[1] TRUE
Esto debería dar un valor booleano TRUE ya que 109.25 es mayor que 102
Limpia tu entorno de trabajo borrando las variables de mass y age.
Podemos usar el comando rm para realizar esta tarea:
rm(age, mass)- ¿Cómo puedo gestionar mis proyectos en R?
- Crear proyectos independientes en RStudio
Introducción
El proceso científico es naturalmente incremental, y la vida de muchos proyectos comienza como notas aleatorias, algún código, luego un manuscrito, y eventualmente todo está mezclado.
La mayoría de la gente tiende a organizar sus proyectos de esta manera:
Hay muchas razones de por qué debemos siempre evitar esto:
-
Es realmente difícil saber cuál versión de tus datos es la original y cuál es la modificada.
-
Es muy complicado porque se mezclan archivos con varias extensiones juntas.
-
Probablemente te lleve mucho tiempo encontrar realmente cosas, y relacionar las figuras correctas con el código exacto que ha sido utilizado para generarlas.
Un buen diseño del proyecto finalmente hará tu vida más fácil:
-
Ayudará a garantizar la integridad de tus datos.
-
Hace que sea más simple compartir tu código con alguien más (un compañero de laboratorio, colaborador o supervisor).
-
Permite cargar fácilmente tu código junto con el envío de tu manuscrito.
-
Hace que sea más fácil retomar un proyecto después de un descanso.
Afortunadamente hay herramientas y paquetes que pueden ayudarte a gestionar tu trabajo con efectividad.
Uno de los aspectos más poderosos y útiles de RStudio es su funcionalidad de gestión de proyectos. Lo utilizaremos hoy para crear un proyecto autocontenido y reproducible.
Vamos a crear un proyecto en RStudio:
-
Hacer clic en el menú “File”, luego en “New Project”.
-
Hacer clic en “New Directory”.
-
Hacer clic en “Empty Project”.
-
Introducir el nombre del directorio para guardar tu proyecto, por ejemplo: “my_project”.
-
Si está disponible, seleccionar la casilla de verificación “Create a git repository.”
-
Hacer clic en el botón “Create Project”.
Ahora cuando inicies R en este directorio de proyectos, o abras este proyecto con RStudio, todo nuestro trabajo estará completamente autocontenido en este directorio.
Aunque no existe una “mejor” forma de diseñar un proyecto, existen algunos principios generales que deben cumplirse para facilitar su gestión:
Este es probablemente el objetivo más importante al configurar un proyecto. Los datos suelen consumir mucho tiempo y/o ser costosos de recolectar. Trabajar con ellos en un formato en el que pueden ser modificados (por ejemplo, en Excel) significa que nunca estás seguro de donde provienen, o cómo han sido modificados desde su recolección. Por lo tanto, es una buena idea manejar tus datos como de “sólo lectura”.
En muchos casos tus datos estarán “sucios” y necesitarán un preprocesamiento significativo para obtener un formato R (o cualquier otro lenguaje de programación) que te resulte útil. Esta tarea es algunas veces llamada “data munging”. Es útil almacenar estos scripts en una carpeta separada y crear una segunda carpeta de datos de “sólo lectura” para contener los datasets “limpios”.
Todo lo generado por tus scripts debe tratarse como descartable: todo debería poder regenerarse a partir de tus scripts.
Hay muchas diferentes maneras de gestionar esta salida. Es útil tener una carpeta de salida con diferentes subdirectorios para cada análisis por separado. Esto hace que sea más fácil después, ya que muchos de nuestros análisis son exploratorios y no terminan siendo utilizados en el proyecto final, y algunos de los análisis se comparten entre proyectos.
Good Enough Practices for Scientific Computing brinda las siguientes recomendaciones para la organización de proyectos:
-
Coloque cada proyecto en su propio directorio, el cual lleva el nombre del proyecto.
-
Coloque documentos de texto asociados con proyecto en el directorio
doc. -
Coloque los datos sin procesar y los metadatos en el directorio
data, y archivos generados durante la limpieza y análisis en el directorioresults. -
Coloque los scripts fuente del proyecto y los programas en el directorio
src, y programas traídos de otra parte o compilados localmente en el directoriobin. -
Nombre todos archivos de tal manera que reflejen su contenido o función.
Una manera de automatizar la gestión de un proyecto es instalar el paquete ProjectTemplate. Este paquete configurará una estructura de directorios ideal para la gestión de proyectos. Esto es muy útil ya que te permite tener tu pipeline/workflow de análisis organizado y estructurado. Junto con la funcionalidad predeterminada del proyecto RStudio y Git, podrás realizar el seguimiento de tu trabajo y compartirlo con colaboradores.
-
Instala
ProjectTemplate. -
Carga la librería.
-
Inicializa el proyecto:
install.packages("ProjectTemplate")
library("ProjectTemplate")
create.project("../my_project", merge.strategy = "allow.non.conflict")Para más información de ProjectTemplate y su functionalidad visita la página ProjectTemplate.
Una de las maneras más efectivas de trabajar con R es comenzar escribiendo el código que deseas que se ejecute directamente en un script .R, y enseguida ejecutar las líneas seleccionadas (ya sea utilizando los atajos del teclado en RStudio o haciendo clic en el botón “Run”) en la consola interactiva de R.
Cuando tu proyecto se encuentra en sus primeras etapas, el archivo script inicial .R generalmente contendrá muchas líneas de código ejecutadas directamente. Conforme vaya madurando, fragmentos reutilizables podrán ser llevados a sus propias funciones. Es buena idea separar estas funciones en dos carpetas separadas; una para guardar funciones útiles que reutilizarás a través del análisis y proyectos, y una para guardar los scripts de análisis.
Puedes encontrarte utilizando datos o scripts de análisis a través de varios proyectos. Normalmente, deseas evitar la duplicación para ahorrar espacio y evitar actualizar el código en múltiples lugares.
En este caso, es útil hacer “links simbólicos”, los cuales son esencialmente accesos directos a archivos en otro lugar en un sistema de archivos. En Linux y OS X puedes utilizar el comando ln -s, y en Windows crear un acceso directo o utilizar el comando mklink desde la terminal de Windows.
Ahora que tenemos una buena estructura de directorios colocaremos/guardaremos los archivos de datos en el directorio data/.
Descargar los datos gapminder de aquí.
-
Descargar el archivo (CTRL + S, clic botón derecho del ratón -> “Guardar como…”, o Archivo -> “Guardar página como…”)
-
Asegúrate de que esté guardado con el nombre
gapminder-FiveYearData.csv -
Guardar el archivo en la carpeta
data/dentro de tu proyecto.
Más delante cargaremos e inspeccionaremos estos datos.
Es útil tener una idea general sobre el dataset, directamente desde la línea de comandos, antes de cargarlo en R. Comprender mejor el dataset será útil al tomar decisiones sobre cómo cargarlo en R. Utiliza la terminal de línea de comandos para contestar las siguientes preguntas:
-
¿Cuál es el tamaño del archivo?
-
¿Cuántas líneas de datos contiene?
-
¿Cuáles tipos de valores están almacenados en este archivo?
Al ejecutar estos comandos en la terminal:
ls -lh data/gapminder-FiveYearData.csv-rw-r--r-- 1 CRISTIAN FARIAS 80K Oct 8 13:27 data/gapminder-FiveYearData.csv
El tamaño del archivo es 80K.
wc -l data/gapminder-FiveYearData.csv1705 data/gapminder-FiveYearData.csv
Hay 1705 líneas. Los datos se ven así:
head data/gapminder-FiveYearData.csvcountry,year,pop,continent,lifeExp,gdpPercap
Afghanistan,1952,8425333,Asia,28.801,779.4453145
Afghanistan,1957,9240934,Asia,30.332,820.8530296
Afghanistan,1962,10267083,Asia,31.997,853.10071
Afghanistan,1967,11537966,Asia,34.02,836.1971382
Afghanistan,1972,13079460,Asia,36.088,739.9811058
Afghanistan,1977,14880372,Asia,38.438,786.11336
Afghanistan,1982,12881816,Asia,39.854,978.0114388
Afghanistan,1987,13867957,Asia,40.822,852.3959448
Afghanistan,1992,16317921,Asia,41.674,649.3413952
Puedes abrir rápidamente una terminal en RStudio usando la opción del menú Tools -> Shell….
Es importante llevar a cabo el control de versiones en un proyecto. Ve aquí para una buena lección donde se describe el uso de Git con R Studio.
-
Usar RStudio para crear y gestionar proyectos con un diseño consistente.
-
Tratar los datos brutos como de sólo lectura.
-
Tratar la salida generada como disponible.
-
Definición y aplicación de funciones separadas.
- ¿Cómo puedo obtener ayuda en R?
-
Poder leer archivos de ayuda de R, para funciones y operadores especiales
-
Poder usar vistas de tareas CRAN para identificar paquetes para resolver un problema
-
Para poder buscar ayuda de tus compañeros
Palabras clave
Comando : Traducción
help : ayuda
vignette : viñeta
R, y cada paquete, proporciona archivos de ayuda para las funciones. La sintaxis general para buscar ayuda en cualquier función, “function_name”, de una función específica que esté en un paquete cargado dentro de tu namespace (tu sesión interactiva en R):
?function_name
help(function_name)Esto cargará una página de ayuda en RStudio (o como texto sin formato en R por sí mismo).
Cada página de ayuda se divide en secciones:
-
Descripción: una descripción extendida de lo que hace la función.
-
Uso: los argumentos de la función y sus valores predeterminados.
-
Argumentos: una explicación de los datos que espera cada argumento.
-
Detalles: cualquier detalle importante a tener en cuenta.
-
Valor: los datos que regresa la función.
-
Ver también: cualquier función relacionada que pueda serte útil.
-
Ejemplos: algunos ejemplos de cómo usar la función.
Las diferentes funciones pueden tener diferentes secciones, pero estas son las principales que debes tener en cuenta.
Uno de los aspectos más desalentadores de R es la gran cantidad de funciones disponibles. Es muy difícil, si no imposible, recordar el uso correcto para cada función que usas. Afortunadamente, están los archivos de ayuda ¡lo que significa, que no tienes que hacerlo!
Para buscar ayuda en operadores especiales, usa comillas:
?"<-"Muchos paquetes vienen con “viñetas”: tutoriales y documentación de ejemplo extendida. Sin ningún argumento, vignette() listará todas las viñetas disponibles para todos los paquetes instalados; vignette(package="package-name") listará todas las viñetas disponibles para package-name, y vignette("vignette-name") abrirá la viñeta especificada.
Si un paquete no tiene viñetas, generalmente puedes encontrar ayuda escribiendo help("package-name").
Si no estás seguro de en qué paquete está una función, o cómo se escribe específicamente, puedes hacer una búsqueda difusa:
??function_nameSi no sabes qué función o paquete necesitas usar, utiliza CRAN Task Views es una lista especialmente mantenida de paquetes agrupados en campos. Este puede ser un buen punto de partida.
Si tienes problemas para usar una función, 9 de cada 10 veces, las respuestas que estas buscando ya han sido respondidas en Stack Overflow. Puedes buscar usando la etiqueta [r].
Si no puedes encontrar la respuesta, hay algunas funciones útiles para ayudarte a hacer una pregunta a tus compañeros:
?dputDescargará los datos con los que estás trabajando en un formato para que puedan ser copiados y pegados por cualquier otra persona en su sesión de R.
sessionInfo()R version 4.4.1 (2024-06-14 ucrt)
Platform: x86_64-w64-mingw32/x64
Running under: Windows 11 x64 (build 22631)
Matrix products: default
locale:
[1] LC_COLLATE=Spanish_Argentina.utf8
[2] LC_CTYPE=Spanish_Argentina.utf8
[3] LC_MONETARY=Spanish_Argentina.utf8
[4] LC_NUMERIC=C
[5] LC_TIME=Spanish_Argentina.utf8
time zone: America/Buenos_Aires
tzcode source: internal
attached base packages:
[1] stats graphics grDevices utils
[5] datasets methods base
loaded via a namespace (and not attached):
[1] compiler_4.4.1 tools_4.4.1 knitr_1.48
[4] xfun_0.48
Imprimirá tu versión actual de R, así como cualquier paquete que hayas cargado. Esto puede ser útil para otros para ayudar a reproducir y depurar tu problema.
Buscar la ayuda para la función c. ¿Qué tipo de vector crees que crearás si evalúas lo siguiente?:
c(1, 2, 3)
c('d', 'e', 'f')
c(1, 2, 'f')La función c() crea un vector, en el cual todos los elementos son del mismo tipo. En el primer caso, los elementos son numéricos, en el segundo, son character, y en el tercero son character: los valores numéricos son “forzados” para ser characters.
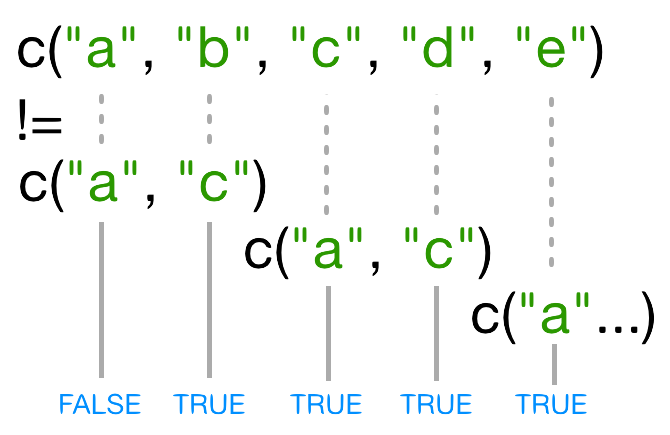
Buscar la ayuda para la función paste. Tendrás que usar esto más tarde. ¿Cuál es la diferencia entre los argumentos sep y collapse?
Busca la ayuda de la función paste(), usa:
help("paste")
?pasteLa diferencia entre sep y collapse es un poco complicada. La función paste acepta cualquier número de argumentos, cada uno de los cuales puede ser un vector de cualquier longitud. El argumento sep especifica la cadena usada entre términos concatenados — por defecto, un espacio. El resultado es un vector tan largo como el argumento más largo proporcionado a paste. En cambio, collapse especifica que después de la concatenación los elementos son colapsados juntos utilizando el separador dado, y el resultado es una sola cadena. e.j.
paste(c("a","b"), "c")[1] "a c" "b c"
paste(c("a","b"), "c", sep = ",")[1] "a,c" "b,c"
paste(c("a","b"), "c", collapse = "|")[1] "a,c|b,c"
paste(c("a","b"), "c", sep = ",", collapse = "|")[1] "a,c|b,c"
(Para más información, ve a la parte inferior de la página de ayuda ?paste y busca los ejemplos, o prueba example('paste').)
Usa la ayuda para encontrar una función (y sus parámetros asociados) que tu puedas usar para cargar datos de un archivo csv en los cuales las columnas están delimitadas con “\ t” (tab) y el punto decimal es un “.” (punto). Esta comprobación para el separador decimal es importante, especialmente si estás trabajando con colegas internacionales ya que diferentes países tienen diferentes convenciones para el punto decimal (i.e. coma vs. punto). sugerencia: usa ??csv para buscar funciones relacionadas con csv.
La función R estándar para leer archivos delimitados por tabuladores con un separador de punto decimal es read.delim(). Tu puedes hacer esto también con read.table(file, sep="\t") (el punto es el separador decimal por defecto para read.table(), aunque es posible que tengas que cambiar también el argumento comment.char si tu archivo de datos contiene caracteres numeral (#)
Otros recursos útiles
- Usar
help()para obtener ayuda online de R.
-
¿Cómo puedo leer datos en R?
-
¿Cuáles son los tipos de datos básicos en R?
-
¿Cómo represento la información categórica en R?
-
Conocer los distintos tipos de datos.
-
Comenzar a explorar los data frames y entender cómo se relacionan con vectors, factors y lists.
-
Ser capaz de preguntar sobre el tipo, clase y estructura de un objeto en R.
-
Conocer y entender qué es coerción y cuáles son los distintos tipos de coerciones.
Comando : Significado
data set : conjunto de datos
c : combinar
dim : dimensión
nrow : número de filas
ncol: número de columnas
Una de las características más poderosas de R es su habilidad para manejar datos tabulares - como los que puedes tener en una planilla de cálculo o un archivo CSV. Comencemos creando un dataset llamado gatos que se vea de la siguiente forma:
color,peso,le_gusta_cuerda
mixto,2.1,1
negro,5.0,0
atigrado,3.2,1Podemos usar la función data.frame para crearlo.
gatos <- data.frame(color = c("mixto", "negro", "atigrado"),
peso = c(2.1, 5.0, 3.2),
le_gusta_cuerda = c(1, 0, 1))
gatos color peso le_gusta_cuerda
1 mixto 2.1 1
2 negro 5.0 0
3 atigrado 3.2 1
Crea el archivo data/gatos-data.csv en RStudio usando el ítem del Menú Files -> New folder. Ahi debes crear el directorio data para poder guardar dentro el archivo gatos-data.csv. Ahora sí usa write.csv(gatos, "data/gatos-data.csv", row.names=FALSE) para crear el archivo
Podemos leer el archivo en R con el siguiente comando:
gatos <- read.csv(file = "data/gatos-data.csv", stringsAsFactors = TRUE)
gatos color peso le_gusta_cuerda
1 mixto 2.1 1
2 negro 5.0 0
3 atigrado 3.2 1
La función read.table se usa para leer datos tabulares almacenados en un archivo de texto donde las columnas de datos están separadas por caracteres de puntuación, por ejemplo archivos CSV (csv = valores separados por comas). Las tabulaciones y las comas son los caracteres de puntuación más utilizados para separar o delimitar datos en archivos csv. Para mayor comodidad, R proporciona otras 2 versiones de “read.table”. Estos son: read.csv para archivos donde los datos están separados por comas y read.delim para archivos donde los datos están separados por tabulaciones. De estas tres funciones, read.csv es el más utilizado. Si fuera necesario, es posible anular o modificar el delimitador predeterminado para read.csv y read.delim.
Podemos empezar a explorar el dataset inmediatamente proyectando las columnas usando el operador $:
gatos$peso[1] 2.1 5.0 3.2
gatos$color[1] mixto negro atigrado
Levels: atigrado mixto negro
Podemos hacer otras operaciones sobre las columnas. Por ejemplo, podemos aumentar el peso de todos los gatos con:
gatos$peso + 2[1] 4.1 7.0 5.2
Podemos imprimir los resultados en una oración
paste("El color del gato es", gatos$color)[1] "El color del gato es mixto" "El color del gato es negro"
[3] "El color del gato es atigrado"
Pero qué pasa con:
gatos$peso + gatos$colorWarning in Ops.factor(gatos$peso, gatos$color): '+' not meaningful for factors
[1] NA NA NA
Si adivinaste que el último comando iba a resultar en un error porque 2.1 más "negro" no tiene sentido, estás en lo cierto - y ya tienes alguna intuición sobre un concepto importante en programación que se llama tipos de datos.
No importa cuán complicado sea nuestro análisis, todos los datos en R se interpretan con uno de estos tipos de datos básicos. Este rigor tiene algunas consecuencias importantes.
Hay 5 tipos de datos principales: double, integer, complex, logical and character.
Podemos preguntar cuál es la estructura de datos si usamos la función class:
class(gatos$color)[1] "factor"
class(gatos$peso)[1] "numeric"
También podemos ver que gatos es un data.frame si usamos la función class:
class(gatos)[1] "data.frame"
Para entender mejor este comportamiento, veamos otra de las estructuras de datos en R: el vector.
Un vector en R es esencialmente una lista ordenada de cosas, con la condición especial de que todos los elementos en un vector tienen que ser del mismo tipo de datos básico. Si no eliges un tipo de datos, por defecto R elige el tipo de datos logical. También puedes declarar un vector vacío de cualquier tipo que quieras.
Una indicación del número de elementos en el vector - específicamente los índices del vector, en este caso [1:3] y unos pocos ejemplos de los elementos del vector - en este caso strings vacíos.
Podemos ver que gatos$peso es un vector usando la funcion str.
str(gatos$peso) num [1:3] 2.1 5 3.2
Las columnas de datos que cargamos en data.frames de R son todas vectores y este es el motivo por el cual R requiere que todas las columnas sean del mismo tipo de datos básico.
¿Por qué R es tan obstinado acerca de lo que ponemos en nuestras columnas de datos? ¿Cómo nos ayuda esto?
Al mantener todos los elementos de una columna del mismo tipo, podemos hacer suposiciones simples sobre nuestros datos; si puedes interpretar un elemento en una columna como un número, entonces puedes interpretar todos los elementos como números, y por tanto no hace falta comprobarlo cada vez. Esta consistencia es lo que se suele mencionar como datos limpios; a la larga, la consistencia estricta hace nuestras vidas más fáciles cuando usamos R.
También puedes crear vectores con contenido explícito con la función combine o c():
mi_vector <- c(2,6,3)
mi_vector[1] 2 6 3
str(mi_vector)num [1:3] 2 6 3
Dado lo que aprendimos hasta ahora, ¿qué crees que hace el siguiente código?
otro_vector <- c(2,6,'3')Esto se denomina coerción de tipos de datos y es motivo de muchas sorpresas y la razón por la cual es necesario conocer los tipos de datos básicos y cómo R los interpreta. Cuando R encuentra una mezcla de tipos de datos (en este caso numeric y character) para combinarlos en un vector, va a forzarlos a ser del mismo tipo.
Considera:
vector_coercion <- c('a', TRUE)
str(vector_coercion) chr [1:2] "a" "TRUE"
otro_vector_coercion <- c(0, TRUE)
str(otro_vector_coercion) num [1:2] 0 1
Las reglas de coerción son: logical -> integer -> numeric -> complex -> character, donde -> se puede leer como se transforma en. Puedes intentar forzar la coerción de acuerdo a esta cadena usando las funciones as.:
vector_caracteres <- c('0','2','4')
vector_caracteres[1] "0" "2" "4"
str(vector_caracteres) chr [1:3] "0" "2" "4"
caracteres_coercionados_numerico <- as.numeric(vector_caracteres)
caracteres_coercionados_numerico[1] 0 2 4
numerico_coercionado_logico <- as.logical(caracteres_coercionados_numerico)
numerico_coercionado_logico[1] FALSE TRUE TRUE
Como puedes notar, es sorprendete ver qué pasa cuando R forza la conversión de un tipo de datos a otro. Es decir, si tus datos no lucen como pensabas que deberían lucir, puede ser culpa de la coerción de tipos. Por lo tanto, asegúrate que todos los elementos de tus vectores y las columnas de tus data.frames sean del mismo tipo o te encontrarás con sorpresas desagradables.
Pero la coerción de tipos también puede ser muy útil. Por ejemplo, en los datos de gatos, le_gusta_cuerda es numérica, pero sabemos que los 1s y 0s en realidad representan TRUE y FALSE (una forma habitual de representarlos). Deberíamos usar el tipo de datos logical en este caso, que tiene dos estados: TRUE o FALSE, que es exactamente lo que nuestros datos representan. Podemos convertir esta columna al tipo de datos logical usando la función as.logical:
gatos$le_gusta_cuerda[1] 1 0 1
class(gatos$le_gusta_cuerda)[1] "integer"
gatos$le_gusta_cuerda <- as.logical(gatos$le_gusta_cuerda)
gatos$le_gusta_cuerda[1] TRUE FALSE TRUE
class(gatos$le_gusta_cuerda)[1] "logical"
La función combine, c(), también agregará elementos al final de un vector existente:
ab <- c('a', 'b')
ab[1] "a" "b"
abc <- c(ab, 'c')
abc[1] "a" "b" "c"
También puedes hacer una serie de números así:
mySerie <- 1:5
mySerie[1] 1 2 3 4 5
str(mySerie)int [1:5] 1 2 3 4 5
class(mySerie)[1] "integer"
Finalmente, puedes darle nombres a los elementos de tu vector:
names(mySerie) <- c("a", "b", "c", "d", "e")
mySeriea b c d e
1 2 3 4 5
str(mySerie)Named int [1:5] 1 2 3 4 5
- attr(*, "names")= chr [1:5] "a" "b" "c" "d" ...
class(mySerie)[1] "integer"
Comienza construyendo un vector con los números del 1 al 26. Multiplica el vector por 2 y asigna al vector resultante, los nombres de A hasta Z (Pista: hay un vector pre-definido llamado LETTERS)
x <- 1:26
x <- x * 2
names(x) <- LETTERSOtra estructura de datos importante se llama factor.
str(gatos$color)Factor w/ 3 levels "atigrado","mixto",..: 2 3 1
Los factores usualmente parecen caracteres, pero se usan para representar información categórica. Por ejemplo, construyamos un vector de strings con etiquetas para las coloraciones para todos los gatos en nuestro estudio:
capas <- c('atigrado', 'carey', 'carey', 'negro', 'atigrado')
capas[1] "atigrado" "carey" "carey" "negro" "atigrado"
str(capas)chr [1:5] "atigrado" "carey" "carey" "negro" "atigrado"
Podemos convertir un vector en un factor de la siguiente manera:
categorias <- factor(capas)
class(categorias)[1] "factor"
str(categorias)Factor w/ 3 levels "atigrado","carey",..: 1 2 2 3 1
Ahora R puede interpretar que hay tres posibles categorías en nuestros datos - pero también hizo algo sorprendente: en lugar de imprimir los strings como se las dimos, imprimió una serie de números. R ha reemplazado las categorías con índices numéricos, lo cual es necesario porque muchos cálculos estadísticos usan esa representación para datos categóricos:
class(capas)[1] "character"
class(categorias)[1] "factor"
¿Hay algún factor en nuestro data.frame gatos? ¿Cuál es el nombre? Intenta usar ?read.csv para darte cuenta cómo mantener las columnas de texto como vectores de caracteres en lugar de factores; luego escribe uno o más comandos para mostrar que el factor en gatos es en realidad un vector de caracteres cuando se carga de esta manera.
Una solución es usar el argumento stringAsFactors:
gatos <- read.csv(file="data/gatos-data.csv", stringsAsFactors=FALSE)
str(gatos$color)Otra solución es usar el argumento colClasses que permiten un control más fino.
gatos <- read.csv(file="data/gatos-data.csv", colClasses=c(NA, NA, "character"))
str(gatos$color)Nota: Los nuevos estudiantes encuentran los archivos de ayuda difíciles de entender; asegúrese de hacerles saber que esto es normal, y anímelos a que tomen su mejor opción en función del significado semántico, incluso si no están seguros.
En las funciones de modelado, es importante saber cuáles son los niveles de referencia. Se asume que es el primer factor, pero por defecto los factores están etiquetados en orden alfabetico. Puedes cambiar esto especificando los niveles:
misdatos <- c("caso", "control", "control", "caso")
factor_orden <- factor(misdatos, levels = c("control", "caso"))
str(factor_orden) Factor w/ 2 levels "control","caso": 2 1 1 2
En este caso, le hemos dicho explícitamente a R que “control” debería estar representado por 1, y “caso” por 2. Esta designación puede ser muy importante para interpretar los resultados de modelos estadísticos!
Otra estructura de datos que querrás en tu bolsa de trucos es list. Una lista es más simple en algunos aspectos que los otros tipos, porque puedes poner cualquier cosa que tú quieras en ella:
lista <- list(1, "a", TRUE, 1+4i)
lista[[1]]
[1] 1
[[2]]
[1] "a"
[[3]]
[1] TRUE
[[4]]
[1] 1+4i
otra_lista <- list(title = "Numbers", numbers = 1:10, data = TRUE )
otra_lista$title
[1] "Numbers"
$numbers
[1] 1 2 3 4 5 6 7 8 9 10
$data
[1] TRUE
Ahora veamos algo interesante acerca de nuestro data.frame; ¿Qué pasa si corremos la siguiente línea?
typeof(gatos)[1] "list"
Vemos que los data.frames parecen listas ‘en su cara oculta’ - esto es porque un data.frame es realmente una lista de vectores y factores, como debe ser - para mantener esas columnas que son una combinación de vectores y factores, el data.frame necesita algo más flexible que un vector para poner todas las columnas juntas en una tabla. En otras palabras, un data.frame es una lista especial en la que todos los vectores deben tener la misma longitud.
En nuestro ejemplo de gatos, tenemos una variable integer, una double y una logical. Como ya hemos visto, cada columna del data.frame es un vector.
gatos$color[1] mixto negro atigrado
Levels: atigrado mixto negro
gatos[,1][1] mixto negro atigrado
Levels: atigrado mixto negro
typeof(gatos[,1])[1] "integer"
str(gatos[,1])Factor w/ 3 levels "atigrado","mixto",..: 2 3 1
Cada fila es una observación de diferentes variables del mismo data.frame, y por lo tanto puede estar compuesto de elementos de diferentes tipos.
gatos[1,] color peso le_gusta_cuerda
1 mixto 2.1 TRUE
typeof(gatos[1,])[1] "list"
str(gatos[1,])'data.frame': 1 obs. of 3 variables:
$ color : Factor w/ 3 levels "atigrado","mixto",..: 2
$ peso : num 2.1
$ le_gusta_cuerda: logi TRUE
Hay varias maneras sutilmente diferentes de indicar variables, observaciones y elementos de data.frames:
-
gatos[1] -
gatos[[1]] -
gatos$color -
gatos["color"] -
gatos[1, 1] -
gatos[, 1] -
gatos[1, ]
Investiga cada uno de los ejemplos anteriores y explica el resultado de cada uno.
Sugerencia: Usa la función typeof() para examinar el resultado en cada caso.
gatos[1] color
1 mixto
2 negro
3 atigrado
Podemos interpretar un data frame como una lista de vectores. Un único par de corchetes [1] resulta en la primera proyección de la lista, como otra lista. En este caso es la primera columna del data frame.
gatos[[1]][1] mixto negro atigrado
Levels: atigrado mixto negro
El doble corchete [[1]] devuelve el contenido del elemento de la lista. En este caso, es el contenido de la primera columna, un vector de tipo factor.
gatos$color[1] mixto negro atigrado
Levels: atigrado mixto negro
Este ejemplo usa el caracter $ para direccionar elementos por nombre. color es la primera columna del data frame, de nuevo un vector de tipo factor.
gatos["color"] color
1 mixto
2 negro
3 atigrado
Aquí estamos usando un solo corchete ["color"] reemplazando el número del índice con el nombre de la columna. Como el ejemplo 1, el objeto devuelto es un list.
gatos[1, 1][1] mixto
Levels: atigrado mixto negro
Este ejemplo usa un solo corchete, pero esta vez proporcionamos coordenadas de fila y columna. El objeto devuelto es el valor en la fila 1, columna 1. El objeto es un integer pero como es parte de un vector de tipo factor, R muestra la etiqueta “mixto” asociada con el valor entero.
gatos[, 1][1] mixto negro atigrado
Levels: atigrado mixto negro
Al igual que en el ejemplo anterior, utilizamos corchetes simples y proporcionamos las coordenadas de fila y columna. La coordenada de la fila no se especifica, R interpreta este valor faltante como todos los elementos en este column vector.
gatos[1, ] color peso le_gusta_cuerda
1 mixto 2.1 TRUE
De nuevo, utilizamos el corchete simple con las coordenadas de fila y columna. La coordenada de la columna no está especificada. El valor de retorno es una list que contiene todos los valores en la primera fila.
Por último, pero no menos importante, están las matrices. Podemos declarar una matriz llena de ceros de la siguiente forma:
matrix_example <- matrix(0, ncol=6, nrow=3)
matrix_example [,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0 0 0 0 0 0
[2,] 0 0 0 0 0 0
[3,] 0 0 0 0 0 0
Y de manera similar a otras estructuras de datos, podemos preguntar cosas sobre la matriz:
class(matrix_example)[1] "matrix" "array"
typeof(matrix_example)[1] "double"
str(matrix_example)num [1:3, 1:6] 0 0 0 0 0 0 0 0 0 0 ...
dim(matrix_example)[1] 3 6
nrow(matrix_example)[1] 3
ncol(matrix_example)[1] 6
¿Cuál crees que es el resultado del comando length(matrix_example)? Inténtalo. ¿Estabas en lo correcto? ¿Por qué / por qué no?
¿Cuál crees que es el resultado del comando length(matrix_example)?
matrix_example <- matrix(0, ncol=6, nrow=3)
length(matrix_example)[1] 18
Debido a que una matriz es un vector con atributos de dimensión añadidos, length proporciona la cantidad total de elementos en la matriz.
Construye otra matriz, esta vez conteniendo los números 1:50, con 5 columnas y 10 renglones. ¿Cómo llenó la función matrix de manera predeterminada la matriz, por columna o por renglón? Investiga como cambiar este comportamento. (Sugerencia: lee la documentación de la función matrix.)
documentación de la función matrix.)
Construye otra matriz, esta vez conteniendo los números 1:50, con 5 columnas y 10 renglones. ¿Cómo llenó la función matrix de manera predeterminada la matriz, por columna o por renglón? Investiga como cambiar este comportamento. (Sugerencia: lee la documentación de la función matrix.)
x <- matrix(1:50, ncol=5, nrow=10)
x <- matrix(1:50, ncol=5, nrow=10, byrow = TRUE) # to fill by rowCrea una lista de longitud dos que contenga un vector de caracteres para cada una de las secciones en esta parte del curso:
-
tipos de datos
-
estructura de datos
Inicializa cada vector de caracteres con los nombres de los tipos de datos y estructuras de datos que hemos visto hasta ahora.
dataTypes <- c('double', 'complex', 'integer', 'character', 'logical')
dataStructures <- c('data.frame', 'vector', 'factor', 'list', 'matrix')
answer <- list(dataTypes, dataStructures)Nota: es útil hacer una lista en el pizarrón o en papel colgado en la pared listando todos los tipos y estructuras de datos y mantener la lista durante el resto del curso para recordar la importancia de estos elementos básicos.
Considera la salida de R para la siguiente matriz:
[,1] [,2]
[1,] 4 1
[2,] 9 5
[3,] 10 7
¿Cuál fué el comando correcto para escribir esta matriz? Examina cada comando e intenta determinar el correcto antes de escribirlos. Piensa en qué matrices producirán los otros comandos.
-
matrix(c(4, 1, 9, 5, 10, 7), nrow = 3) -
matrix(c(4, 9, 10, 1, 5, 7), ncol = 2, byrow = TRUE) -
matrix(c(4, 9, 10, 1, 5, 7), nrow = 2) -
matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)
Considera la salida de R para la siguiente matriz:
[,1] [,2]
[1,] 4 1
[2,] 9 5
[3,] 10 7
¿Cuál era el comando correcto para escribir esta matriz? Examina cada comando e intenta determinar el correcto antes de escribirlos. Piensa en qué matrices producirán los otros comandos.
matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)-
Usar
read.csvpara leer los datos tabulares en R. -
Los vectores, factores, listas y dataframes son estructuras de datos en R
-
Los tipos de datos básicos en R son double, integer, complex, logical, y character.
-
Los factors representan variables categóricas en R.
- ¿Cómo puedo manipular un data frame?
-
Poder agregar y quitar filas y columnas.
-
Poder quitar filas con valores
NA. -
Poder anexar dos data frames.
-
Poder articular qué es un
factory cómo convertir entrefactorycharacter. -
Poder entender las propiedades básicas de un data frame, incluyendo tamaño, clase o tipo de columnas, nombres y primeras filas.
A esta altura, ya viste los tipos y estructuras de datos básicos de R y todo lo que hagas va a ser una manipulación de esas herramientas. Ahora pasaremos a aprender un par de cosas sobre cómo trabajar con la clase data frame (la estructura de datos que usarás la mayoría del tiempo y que será la estrella del show). Un data frame es la tabla que creamos al cargar información de un archivo csv.
Comando : Traducción
nrow: número de filas
ncol: número de columnas
rbind: combinar filas
cbind: combinar columnas
Aprendimos que las columnas en un data frame son vectores. Por lo tanto, sabemos que nuestros datos son consistentes con el tipo de dato dentro de esa columna. Si queremos agregar una nueva columna, podemos empezar por crear un nuevo vector:
gatos color peso legusta_la_cuerda
1 mixto 2.1 1
2 negro 5.0 0
3 atigrado 3.2 1
edad <- c(2,3,5)Podemos entonces agregarlo como una columna via:
cbind(gatos, edad) color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
Tenga en cuenta que fallará si tratamos de agregar un vector con un número diferente de entradas que el número de filas en el marco de datos.
edad <- c(2, 3, 5, 12)
cbind(gatos, edad)Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 4
edad <- c(2, 3)
cbind(gatos, edad)Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 2
¿Por qué no funcionó? Claro, R quiere ver un elemento en nuestra nueva columna para cada fila de la tabla:
Para que funcione, debemos tener nrow(gatos) = length(edad). Vamos a sobrescribir el contenido de los gatos con nuestro nuevo marco de datos.
edad <- c(2, 3, 5)
gatos <- cbind(gatos, edad)
gatos color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
Ahora, qué tal si agregamos filas, en este caso, la última vez vimos que las filas de un data frame están compuestas por listas:
nueva_fila <- list("carey", 3.3, TRUE, 9)
gatos <- rbind(gatos, nueva_fila)Warning in `[<-.factor`(`*tmp*`, ri, value = "carey"): invalid factor level, NA
generated
Qué significa el error que nos da R? ‘invalid factor level’ nos dice algo acerca de factores (factors)… pero qué es un factor? Un factor es un tipo de datos en R. Un factor es una categoría (por ejemplo, color) con la que R puede hacer ciertas operaciones. Por ejemplo:
colores <- factor(c("negro","canela","canela","negro"))
levels(colores)[1] "canela" "negro"
nlevels(colores)[1] 2
Se puede reorganizar el orden de los factores para que en lugar de que aparezcan por orden alfabético sigan el orden elegido por el usuario.
colores ## el orden actual[1] negro canela canela negro
Levels: canela negro
colores <- factor(colores, levels = c("negro", "canela"))
colores # despues de re-organizar[1] negro canela canela negro
Levels: negro canela
Los objetos de la clase factor son otro tipo de datos que debemos usar con cuidado. Cuando R crea un factor, únicamente permite los valores que originalmente estaban allí cuando cargamos los datos. Por ejemplo, en nuestro caso ‘negro’, ‘canela’ y ‘atigrado’. Cualquier categoría nueva que no entre en esas categorías será rechazada (y se conviertirá en NA).
La advertencia (Warning) nos está diciendo que agregamos ‘carey’ a nuestro factor color. Pero los otros valores, 3.3 (de tipo numeric), TRUE (de tipo logical), y 9 (de tipo numeric) se añadieron exitosamente a peso, le_gusta_cuerda, y edad, respectivamente, dado que esos valores no son de tipo factor. Para añadir una nueva categoría ‘carey’ al data frame gatos en la columna color, debemos agregar explícitamente a ‘carey’ como un nuevo nivel (level) en el factor:
levels(gatos$color)[1] "atigrado" "mixto" "negro"
levels(gatos$color) <- c(levels(gatos$color), 'carey')
gatos <- rbind(gatos, list("carey", 3.3, TRUE, 9))De manera alternativa, podemos cambiar la columna a tipo character. En este caso, perdemos las categorías, pero a partir de ahora podemos incorporar cualquier palabra a la columna, sin problemas con los niveles del factor.
str(gatos)'data.frame': 5 obs. of 4 variables:
$ color : Factor w/ 4 levels "atigrado","mixto",..: 2 3 1 NA 4
$ peso : num 2.1 5 3.2 3.3 3.3
$ legusta_la_cuerda: num 1 0 1 1 1
$ edad : num 2 3 5 9 9
gatos$color <- as.character(gatos$color)
str(gatos)'data.frame': 5 obs. of 4 variables:
$ color : chr "mixto" "negro" "atigrado" NA ...
$ peso : num 2.1 5 3.2 3.3 3.3
$ legusta_la_cuerda: num 1 0 1 1 1
$ edad : num 2 3 5 9 9
Imaginemos que, como los perros, 1 año humano es equivalente a 7 años en los gatos (La compañía Purina usa un algoritmo más sofisticado).
-
Crea un vector llamado
human.edadmultiplicandogatos$edadpor 7. -
Convierte
human.edada factor. -
Convierte
human.edadde nuevo a un vector numérico usando la funciónas.numeric(). Ahora, divide por 7 para regresar a las edades originales. Explica lo sucedido.
-
human.edad <- gatos$edad * 7 -
human.edad <- factor(human.edad)oas.factor(human.edad)las dos opciones funcionan igual de bien. -
as.numeric(human.edad)produce1 2 3 4 4porque los factores se guardan como objetos de tipo entero integer (1:4), cada uno de los cuales tiene asociado una etiqueta label (28, 35, 56, y 63). Convertir un objeto de un tipo de datos a otro, por ejemplo de factor a numeric nos dá los enteros, no las etiquetas labels. Si queremos los números originales, necesitamos un paso intermedio, debemos convertirhuman.edadal tipo character y luego a numeric (¿cómo funciona esto?). Esto aparece en la vida real cuando accidentalmente incluimos un character en alguna columna de nuestro archivo .csv, que se suponía que únicamente contendría números. Tendremos este problema, si al leer el archivo olvidamos incluirstringsAsFactors=FALSE.
Ahora sabemos cómo agregar filas y columnas a nuestro data frame en R, pero en nuestro primer intento para agregar un gato llamado ‘carey’ agregamos una fila que no sirve.
gatos color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
4 <NA> 3.3 1 9
5 carey 3.3 1 9
Podemos pedir el data frame sin la fila errónea:
gatos[-4,] color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
5 carey 3.3 1 9
Notar que -4 significa que queremos remover la cuarta fila, la coma sin nada detrás indica que se aplica a todas las columnas. Podríamos remover ambas filas en un llamado usando ambos números dentro de un vector: gatos[c(-4,-5),]
Alternativamente, podemos eliminar filas que contengan valores NA:
na.omit(gatos) color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
5 carey 3.3 1 9
Volvamos a asignar el nuevo resultado output al data frame gatos, así nuestros cambios son permanentes:
gatos <- na.omit(gatos)También podemos eliminar columnas en un data frame. Hay dos formas de eliminar una columna: por número o nombre de índice.
gatos[,-4] color peso legusta_la_cuerda
1 mixto 2.1 1
2 negro 5.0 0
3 atigrado 3.2 1
5 carey 3.3 1
Observa la coma sin nada antes, lo que indica que queremos mantener todas las filas.
Alternativamente, podemos quitar la columna usando el nombre del índice.
drop <- names(gatos) %in% c("edad")
gatos[,!drop] color peso legusta_la_cuerda
1 mixto 2.1 1
2 negro 5.0 0
3 atigrado 3.2 1
5 carey 3.3 1
La clave que hay que recordar al añadir datos a un data frame es que las columnas son vectores o factores, mientras que las filas son listas. Podemos pegar dos data frames usando rbind que significa unir las filas (verticalmente):
gatos <- rbind(gatos, gatos)
gatos color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
5 carey 3.3 1 9
11 mixto 2.1 1 2
21 negro 5.0 0 3
31 atigrado 3.2 1 5
51 carey 3.3 1 9
Pero ahora los nombres de las filas rownames son complicados. Podemos removerlos y R los nombrará nuevamente, de manera secuencial:
rownames(gatos) <- NULL
gatos color peso legusta_la_cuerda edad
1 mixto 2.1 1 2
2 negro 5.0 0 3
3 atigrado 3.2 1 5
4 carey 3.3 1 9
5 mixto 2.1 1 2
6 negro 5.0 0 3
7 atigrado 3.2 1 5
8 carey 3.3 1 9
Puedes crear un nuevo data frame desde R con la siguiente sintaxis:
df <- data.frame(id = c('a', 'b', 'c'),
x = 1:3,
y = c(TRUE, TRUE, FALSE),
stringsAsFactors = FALSE)Crear un data frame que contenga la siguiente información personal:
-
nombre
-
apellido
-
número favorito
Luego usa rbind para agregar una entrada para la gente sentada alrededor tuyo. Finalmente, usa cbind para agregar una columna con espacio para que cada persona conteste a la siguiente pregunta: “¿Es hora de una pausa?”
df <- data.frame(first = c('Grace'),
apellido = c('Hopper'),
numero_favorito = c(0),
stringsAsFactors = FALSE)
df <- rbind(df, list('Marie', 'Curie', 238) )
df <- cbind(df, cafe = c(TRUE,TRUE))Hasta ahora, hemos visto las manipulaciones básicas que pueden hacerse en un data frame. Ahora, vamos a extender esas habilidades con un ejemplo más real. Vamos a importar el gapminder dataset que descargamos previamente:
La función read.table se usa para leer datos tabulares que están guardados en un archivo de texto, donde las columnas de datos están separadas por un signo de puntuación como en los archivos CSV (donde csv es comma-separated values en inglés, es decir, valores separados por comas).
Los signos de puntuación más comunmente usados para separar o delimitar datos en archivos de texto son tabuladores y comas. Por conveniencia, R provee dos versiones de la función read.table. Estas versiones son: read.csv para archivos donde los datos están separados por comas y read.delim para archivos donde los datos están separados por tabuladores. De las tres variantes, read.csv es la más comúnmente usada. De ser necesario, es posible sobrescribir el signo de puntuación usado por defecto para ambas funciones: read.csv y read.delim.
gapminder <- read.csv("data/gapminder-FiveYearData.csv")-
Otro tipo de archivo que puedes encontrar es el separado por tabuladores (.tsv). Para especificar este separador, usa
"\t"oread.delim(). -
Los archivos pueden descargarse de Internet a una carpeta local usando
download.file. La funciónread.csvpuede ser ejecutada para leer el archivo descargado, por ejemplo:
download.file("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/gh-pages/_episodes_rmd/data/gapminder-FiveYearData.csv", destfile = "data/gapminder-FiveYearData.csv")
gapminder <- read.csv("data/gapminder-FiveYearData.csv")- De manera alternativa, puedes leer los archivos directamente en R, usando una dirección web y
read.csv. Es importante notar que, si se hace esto último, no habrá una copia local del archivo csv en tu computadora. Por ejemplo,
gapminder <- read.csv("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/gh-pages/_episodes_rmd/data/gapminder-FiveYearData.csv")- Puedes leer directamente planillas de Excel sin necesidad de convertirlas a texto plano usando el paquete readxl.
Vamos a investigar gapminder un poco; lo primero que hay que hacer siempre es ver cómo se ve el dataset usando str:
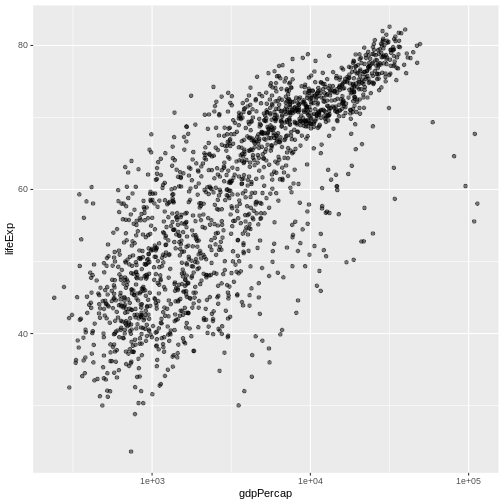
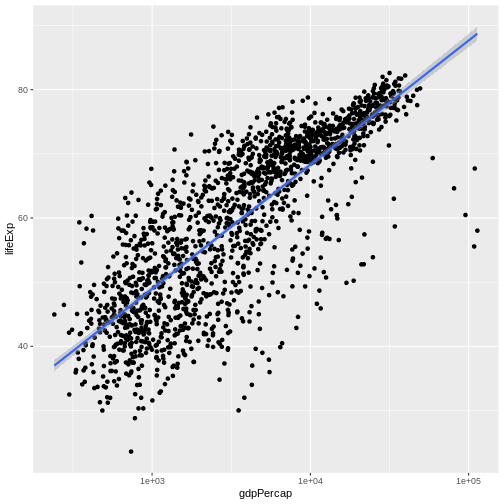
str(gapminder)'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...
También podemos examinar columnas individuales del data frame con la función typeof:
typeof(gapminder$year)[1] "integer"
typeof(gapminder$country)[1] "character"
str(gapminder$country) chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
También podemos interrogar al data frame por la información sobre sus dimensiones; recordando que str(gapminder) dijo que había 1704 observaciones de 6 variables en gapminder, ¿qué piensas que el siguiente código producirá y por qué?
length(gapminder)[1] 6
Un intento certero hubiera sido decir que el largo (length) de un data frame es el número de filas (1704), pero no es el caso; recuerda, un data frame es una lista de vectores y factors.
typeof(gapminder)[1] "list"
Cuando length devuelve 6, es porque gapminder está construida por una lista de 6 columnas. Para conseguir el número de filas, intenta:
nrow(gapminder)[1] 1704
ncol(gapminder)[1] 6
O, para obtener ambos de una vez:
dim(gapminder)[1] 1704 6
Probablemente queremos saber los nombres de las columnas. Para hacerlo, podemos pedir:
colnames(gapminder)[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"
A esta altura, es importante preguntarnos si la estructura de R está en sintonía con nuestra intuición y nuestras expectativas, ¿tienen sentido los tipos de datos reportados para cada columna? Si no lo tienen, necesitamos resolver cualquier problema antes de que se conviertan en sorpresas ingratas luego. Podemos hacerlo usando lo que aprendimos sobre cómo R interpreta los datos y la importancia de la estricta consistencia con la que registramos los datos.
Una vez que estamos contentos con el tipo de datos y que la estructura parece razonable, es tiempo de empezar a investigar nuestros datos. Mira las siguientes líneas:
head(gapminder) country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134
También es útil revisar algunas líneas en el medio y el final del data frame ¿Cómo harías eso?
Buscar líneas exactamente en el medio no es tan difícil, pero simplemente revisar algunas lineas al azar es suficiente. ¿cómo harías eso?
Para revisar las últimas líneas del data frame R tiene una función para esto:
tail(gapminder) country year pop continent lifeExp gdpPercap
1699 Zimbabwe 1982 7636524 Africa 60.363 788.8550
1700 Zimbabwe 1987 9216418 Africa 62.351 706.1573
1701 Zimbabwe 1992 10704340 Africa 60.377 693.4208
1702 Zimbabwe 1997 11404948 Africa 46.809 792.4500
1703 Zimbabwe 2002 11926563 Africa 39.989 672.0386
1704 Zimbabwe 2007 12311143 Africa 43.487 469.7093
tail(gapminder, n = 15) country year pop continent lifeExp gdpPercap
1690 Zambia 1997 9417789 Africa 40.238 1071.3538
1691 Zambia 2002 10595811 Africa 39.193 1071.6139
1692 Zambia 2007 11746035 Africa 42.384 1271.2116
1693 Zimbabwe 1952 3080907 Africa 48.451 406.8841
1694 Zimbabwe 1957 3646340 Africa 50.469 518.7643
1695 Zimbabwe 1962 4277736 Africa 52.358 527.2722
1696 Zimbabwe 1967 4995432 Africa 53.995 569.7951
1697 Zimbabwe 1972 5861135 Africa 55.635 799.3622
1698 Zimbabwe 1977 6642107 Africa 57.674 685.5877
1699 Zimbabwe 1982 7636524 Africa 60.363 788.8550
1700 Zimbabwe 1987 9216418 Africa 62.351 706.1573
1701 Zimbabwe 1992 10704340 Africa 60.377 693.4208
1702 Zimbabwe 1997 11404948 Africa 46.809 792.4500
1703 Zimbabwe 2002 11926563 Africa 39.989 672.0386
1704 Zimbabwe 2007 12311143 Africa 43.487 469.7093
Para revisar algunas lineas al azar?
La solución que presentamos aquí utiliza funciones anidadas, por ejemplo una función es el argumento de otra función. Esto te puede parecer nuevo, pero ya lo haz usado. Recuerda my_dataframe[rows, cols] imprime el data frame con la sección de filas y columnas definidas (incluso puedes seleccionar un rando de filas y columnas usando : por ejemplo). Para obtener un número al azar o varios números al azar R tiene una función llamada sample.
gapminder[sample(nrow(gapminder), 5), ] country year pop continent lifeExp gdpPercap
1090 Netherlands 1997 15604464 Europe 78.030 30246.131
672 Hong Kong China 2007 6980412 Asia 82.208 39724.979
1392 Slovenia 2007 2009245 Europe 77.926 25768.258
1131 Nigeria 1962 41871351 Africa 39.360 1150.927
1129 Nigeria 1952 33119096 Africa 36.324 1077.282
Para que nuestro análisis sea reproducible debemos poner el código en un script al que podremos volver y editar en el futuro.
Ve a Archivo -> nuevo -> R script, y crea un script de R llamado load-gapminder.R para cargar el dataset gapminder. Ponlo en el directorio scripts/ y agrégalo al control de versiones.
Ejecuta el script usando la función source, usando el path como su argumento o apretando el botón de “source” en RStudio.
Los contenidos de scripts/load-gapminder.R:
download.file("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/gh-pages/_episodes_rmd/data/gapminder-FiveYearData.csv", destfile = "data/gapminder-FiveYearData.csv")
gapminder <- read.csv(file = "data/gapminder-FiveYearData.csv")Para ejecutar el script y cargar los archivos en la variable gapminder:
source(file = "scripts/load-gapminder.R")Leer el output de str(gapminder) de nuevo; esta vez, usar lo que has aprendido de factores, listas y vectores, las funciones como colnames y dim para explicar qué significa el output de str. Si hay partes que no puedes entender, discútelo con tus compañero
El objeto gapminder es un data frame con columnas
-
countryycontinentcomo factors. -
yearcomo integer vector. -
pop,lifeExp, andgdpPercapcomo numeric vectors.
-
Usar
cbind()para agregar una nueva columna a un data frame -
Usar
rbind()para agregar una nueva fila a un data frame -
Quitar filas de un data frame
-
Usar
na.omit()para remover filas de un data frame con valoresNA -
Usar
levels()yas.character()para explorar y manipular columnas de clase factor -
Usar
str(),nrow(),ncol(),dim(),colnames(),rownames(),head()ytypeof()para entender la estructura de un data frame -
Leer un archivo csv usando
read.csv() -
Entender el uso de
length()en un data frame
- ¿Cómo puedo trabajar con subconjuntos de datos en R?
-
Ser capaz de hacer subconjuntos de vectores, factores, matrices, listas y data frames.
-
Ser capaz de extraer uno o multiples elementos: por posición, por nombre, o usando operaciones de comparación.
-
Ser capaz de saltar y quitar elementos de diferentes estructuras de datos.
R dispone de muchas operaciones para generar subconjuntos. Dominarlas te permitirá hacer fácilmente operaciones muy complejas en cualquier dataset.
Existen seis maneras distintas por las cuales se puede hacer un subconjunto de datos de cualquier objeto, y existen tres operadores distintos para hacer subconjuntos para las diferentes estructuras de datos.
Empecemos con el caballito de batalla de R: un vector numérico.
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
x a b c d e
5.4 6.2 7.1 4.8 7.5
En R, un vector puede contener palabras, números o valores lógicos. Estos son llamados vectores atómicos ya que no se pueden simplificar más.
Ya que creamos un vector ejemplo juguemos con él, ¿cómo podemos acceder a su contenido?
Para extraer elementos o datos de un vector podemos usar su índice correspondiente, empezando por uno:
x[1] a
5.4
x[4] d
4.8
No lo parece, pero el operador corchetes es una función. Para los vectores (y las matrices), esto significa “dame el n-ésimo elemento”.
También podemos pedir varios elementos al mismo tiempo:
x[c(1, 3)] a c
5.4 7.1
O podemos tomar un rango del vector:
x[1:4] a b c d
5.4 6.2 7.1 4.8
el operador : crea una sucesión de números del valor a la izquierda hasta el de la derecha.
1:4[1] 1 2 3 4
c(1, 2, 3, 4)[1] 1 2 3 4
También podemos pedir el mismo elemento varias veces:
x[c(1,1,3)] a a c
5.4 5.4 7.1
Si pedimos por índices mayores a la longitud del vector, R regresará un valor faltante.
x[6]<NA>
NA
Este es un vector de longitud uno que contiene un NA, cuyo nombre también es NA.
Si pedimos el elemento en el índice 0, obtendremos un vector vacío.
x[0]named numeric(0)
En varios lenguajes de programación (C y Python por ejemplo), el primer elemento de un vector tiene índice 0. En R, el primer elemento tiene el índice 1.
Si usamos un valor negativo como índice para un vector, R regresará cada elemento excepto lo que se ha especificado:
x[-2] a c d e
5.4 7.1 4.8 7.5
También podemos saltar o no mostrar varios elementos:
x[c(-1, -5)] # o bien x[-c(1,5)] b c d
6.2 7.1 4.8
Un pequeño obstáculo para los novatos ocurre cuando tratan de saltar rangos de elementos de un vector. Es natural tratar de filtrar una sucesión de la siguiente manera:
x[-1:3]Esto nos devuelve un error algo críptico:
Error in x[-1:3]: only 0's may be mixed with negative subscripts
Pero recuerda el orden de las operaciones. : es en realidad una función. Toma como primer elemento -1 y como segundo 3, por lo que se genera la sucesión de números: c(-1, 0, 1, 2, 3).
La solución correcta sería empaquetar la llamada de la función dentro de paréntesis, de está manera el operador - se aplica al resultado:
x[-(1:3)] d e
4.8 7.5
Para quitar los elementos de un vector, será necesario que asignes el resultado de vuelta a la variable:
x <- x[-4]
x a b c e
5.4 6.2 7.1 7.5
Dado el siguiente código:
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
print(x) a b c d e
5.4 6.2 7.1 4.8 7.5
Encuentra al menos 3 comandos distintos que produzcan la siguiente salida:
b c d
6.2 7.1 4.8
Después de encontrar los tres comandos distintos, compáralos con los de tu vecino. ¿Tuvieron distintas estrategias?
x[2:4] b c d
6.2 7.1 4.8
x[-c(1,5)] b c d
6.2 7.1 4.8
x[c("b", "c", "d")] b c d
6.2 7.1 4.8
x[c(2,3,4)] b c d
6.2 7.1 4.8
Podemos extraer elementos usando sus nombres, en lugar de extraerlos por índice:
x <- c(a=5.4, b=6.2, c=7.1, d=4.8, e=7.5) # podemos nombrar un vector en la misma línea
x[c("a", "c")] a c
5.4 7.1
Esta forma es mucho más segura para hacer subconjuntos: las posiciones de muchos elementos pueden cambiar a menudo cuando estamos creando una cadena de subconjuntos, ¡pero los nombres siempre permanecen iguales!
También podemos usar un vector con elementos lógicos para hacer subconjuntos:
x[c(FALSE, FALSE, TRUE, FALSE, TRUE)] c e
7.1 7.5
Dado que los operadores de comparación (e.g. >, <, ==) dan como resultado valores lógicos, podemos usarlos para crear subconjuntos de manera mas sintética: la siguiente instrucción tiene el mismo resultado que el anterior.
x[x > 7] c e
7.1 7.5
Explicando un poco lo que sucedió, la instrucción x>7, genera un vector lógico c(FALSE, FALSE, TRUE, FALSE, TRUE) y después éste selecciona los elementos de x correspondientes a los valores TRUE.
Podemos usar == para imitar el método anterior de indexar con nombre (recordemos que se usa == en vez de = para comparar):
x[names(x) == "a"] a
5.4
Muchas veces queremos combinar varios criterios lógicos. Por ejemplo, tal vez queramos encontrar todos los países en Asia o (en inglés or) Europe y (en inglés and) con esperanza de vida en cierto rango. Existen muchas operaciones para combinar vectores con elementos lógicos en R:
-
&, el operador “lógico AND”: regresaTRUEsi tanto la derecha y la izquierda sonTRUE. -
|, el operador “lógico OR”: regresaTRUE, si la derecha o la izquierda (o ambos) sonTRUE.
A veces encontrarás && y ̣|| en vez de & y |. Los operadores de dos caracteres solo comparan los primeros elementos de cada vector e ignoran las demás elementos. En general no debes usar los operadores de dos caracteres en el análisis de datos; déjalos para la programación, i.e. para decir cuando se ejecutara una instrucción.
!, el operador “lógico NOT”: convierteTRUEaFALSEyFALSEaTRUE. Puede negar una sola condición lógica (e.g.!TRUEse vuelveFALSE), o un vector logical (e.g.!c(TRUE, FALSE)se vuelvec(FALSE, TRUE)).
Más aún, puedes comparar todos los elementos de un vector entre ellos usando la función all (que regresa TRUE si todos los elementos del vector son TRUE) y la función any (que regresa TRUE si uno o más elementos del vector son TRUE).
Dado el siguiente código:
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
print(x) a b c d e
5.4 6.2 7.1 4.8 7.5
Escribe un comando para crear el subconjunto de valores de x que sean mayores a 4 pero menores que 7.
x_subset <- x[x<7 & x>4]
print(x_subset) a b d
5.4 6.2 4.8
Debes tener en cuenta que es posible que múltiples elementos en un vector tengan el mismo nombre. (Para un data frame, las columnas pueden tener el mismo nombre —aunque R intenta evitarlo— pero los nombres de las filas deben ser únicos). Considera estos ejemplos:
x <- 1:3
x[1] 1 2 3
names(x) <- c('a', 'a', 'a')
xa a a
1 2 3
x['a'] # solo devuelve el primer valora
1
x[names(x) == 'a'] # devuelve todos los tres valoresa a a
1 2 3
Recuerda que puedes obtener ayuda para los operadores empaquetándolos entre comillas: help("%in%") o ?"%in%".
Saltarse o eliminar elementos con nombre es un poco más difícil. Si tratamos de omitir un elemento con nombre al negar la cadena, R se queja (de una manera un poco oscura) de que no sabe cómo tomar el valor negativo de una cadena:
x <- c(a=5.4, b=6.2, c=7.1, d=4.8, e=7.5) # comenzamos nuevamente nombrando un vector en la misma línea
x[-"a"]Error in -"a": invalid argument to unary operator
Sin embargo, podemos usar el operador != (no igual) para construir un vector con elementos lógicos, que es lo que nosotros queremos:
x[names(x) != "a"] b c d e
6.2 7.1 4.8 7.5
Saltar varios índices con nombre es un poco más difícil. Supongamos que queremos excluir los elementos "a" y "c", entonces intentamos lo siguiente:
x[names(x)!=c("a","c")]Warning in names(x) != c("a", "c"): longer object length is not a multiple of
shorter object length
b c d e
6.2 7.1 4.8 7.5
R hizo algo, pero también nos lanzó una advertencia que debemos atender -¡y aparentemente nos dió la respuesta incorrecta! (el elemento "c" se encuentra todavía en el vector).
¿Entonces qué hizo el operador != en este caso? Esa es una excelente pregunta.
Tomemos un momento para observar al operador de comparación en este código:
names(x) != c("a", "c")Warning in names(x) != c("a", "c"): longer object length is not a multiple of
shorter object length
[1] FALSE TRUE TRUE TRUE TRUE
¿Por qué R devuelve TRUE como el tercer elemento de este vector, cuando names(x)[3] != "c" es obviamente falso?. Cuando tú usas !=, R trata de comparar cada elemento de la izquierda con el correspondiente elemento de la derecha. ¿Qué pasa cuando tu comparas dos elementos de diferentes longitudes?
Cuando uno de los vectores es más corto que el otro, este se recicla:
En este caso R repite c("a", "c") tantas veces como sea necesario para emparejar names(x), i.e. tenemos c("a","c","a","c","a"). Ya que el valor reciclado "a"no es igual a names(x), el valor de != es TRUE. En este caso, donde el vector de mayor longitud (5) no es múltiplo del más pequeño (2), R lanza esta advertencia. Si hubiéramos sido lo suficientemente desafortunados y names(x) tuviese seis elementos, R silenciosamente hubiera hecho las cosas incorrectas (i.e., no lo que deseábamos hacer). Esta regla de reciclaje puede introducir bugs difíciles de encontrar.
La manera de hacer que R haga lo que en verdad queremos (emparejar cada uno de los elementos del argumento de la izquierda con todos los elementos del argumento de la derecha) es usando el operador %in%. El operador %in% toma cada uno de los elementos del argumento de la izquierda, en este caso los nombres de x, y pegunta, “¿este elemento ocurre en el segundo argumento?” Aquí, como queremos excluir los valores, nosotros también necesitamos el operador ! para cambiar la inclusión por una no inclusión:
x[! names(x) %in% c("a","c") ] b d e
6.2 4.8 7.5
Seleccionar elementos de un vector que empareje con cualquier valor de una lista es una tarea muy común en el análisis de datos. Por ejemplo, el data set de gapminder contiene las variables country y continent, pero no incluye información de la escala. Supongamos que queremos extraer la información de el sureste de Asia: ¿cómo podemos escribir una operación que resulte en un vector lógico que sea TRUE para todos los países en el sureste de Asia y FALSE en otros casos?
Supongamos que se tienen los siguientes datos:
seAsia <- c("Myanmar","Thailand","Cambodia","Vietnam","Laos")
## leer los datos de gapminder que bajamos en el episodio 2
gapminder <- read.csv("data/gapminder-FiveYearData.csv", header=TRUE)
## extraer la columna `country` de la **data frame** (veremos esto luego);
## convertir de factor a caracter;
## y quedarse solo con los elementos no repetidos
countries <- unique(as.character(gapminder$country))Existe una manera incorrecta (usando solamente ==), la cual te dará una advertencia (warning); una manera enredada de hacerlo (usando los operadores lógicos == y |) y una manera elegante (usando %in%). Prueba encontrar esas maneras y explica cómo funcionan (o no ).
-
La manera incorrecta de hacer este problema es
countries==seAsia. Esta lanza una advertencia ("In countries == seAsia : longer object length is not a multiple of shorter object length") y la respuesta incorrecta (un vector con todos los valoresFALSE), ya que ninguno de los valores reciclados deseAsiase emparejaron correctamente para coincidir con los valores decountry. -
La manera enredada (pero técnicamente correcta) de resolver este problema es
(countries=="Myanmar" | countries=="Thailand" |
countries=="Cambodia" | countries == "Vietnam" | countries=="Laos")(o countries==seAsia[1] | countries==seAsia[2] | ...). Esto da los valores correctos, pero esperamos que veas lo raro que se ve (¿qué hubiera pasado si hubiéramos querido seleccionar países de una lista mucho más larga?).
- La mejor manera de resolver este problema es
countries %in% seAsia, la cual es la correcta y la más sencilla de escribir (y leer).
En algún momento encontraremos funciones en R que no pueden manejar valores faltantes, infinito o datos indefinidos.
Existen algunas funciones especiales que puedes usar para filtrar estos datos:
- is.na regresa todas las posiciones de un vector, matrix o data frame que contengan NA (o NaN)
- de la misma manera, is.nan y is.infinite hacen lo mismo para NaN e Inf.
- is.finite regresa todas las posiciones de un vector, matrix o data frame que no contengan NA, NaN o Inf.
- na.omit filtra todos los valores faltantes de un vector.
Habiendo explorado las distintas manera de hacer subconjuntos de vectores, ¿cómo podemos hacer subconjuntos de otras estructuras de datos?
Podemos hacer subconjuntos de factores de la misma manera que con los vectores.
f <- factor(c("a", "a", "b", "c", "c", "d"))
f[f == "a"][1] a a
Levels: a b c d
f[f %in% c("b", "c")][1] b c c
Levels: a b c d
f[1:3][1] a a b
Levels: a b c d
Saltar elementos no quita el nivel, incluso cuando no existan datos en esa categoría del factor:
f[-3][1] a a c c d
Levels: a b c d
También podemos hacer subconjuntos de matrices usando la función [ En este caso toma dos argumentos: el primero se aplica a las filas y el segundo a las columnas:
set.seed(1)
m <- matrix(rnorm(6*4), ncol=4, nrow=6)
m[3:4, c(3,1)] [,1] [,2]
[1,] 1.12493092 -0.8356286
[2,] -0.04493361 1.5952808
Siempre puedes dejar el primer o segundo argumento vacío para obtener todas las filas o columnas respectivamente:
m[, c(3,4)] [,1] [,2]
[1,] -0.62124058 0.82122120
[2,] -2.21469989 0.59390132
[3,] 1.12493092 0.91897737
[4,] -0.04493361 0.78213630
[5,] -0.01619026 0.07456498
[6,] 0.94383621 -1.98935170
Si quisieramos acceder a solo una fila o una columna, R automáticamente convertirá el resultado a un vector:
m[3,][1] -0.8356286 0.5757814 1.1249309 0.9189774
Si quieres mantener la salida como una matriz, necesitas especificar un tercer argumento; drop = FALSE:
m[3, , drop=FALSE] [,1] [,2] [,3] [,4]
[1,] -0.8356286 0.5757814 1.124931 0.9189774
A diferencia de los vectores, si tratamos de acceder a una fila o columna fuera de la matriz, R arrojará un error:
m[, c(3,6)]Error in m[, c(3, 6)]: subscript out of bounds
Cuando estamos lidiando con arrays multi-dimensionales, cada uno de los argumentos de [ corresponden a una dimensión. Por ejemplo,en un array 3D, los primeros tres argumentos corresponden a las filas, columnas y profundidad.
Como las matrices son vectores, podemos también hacer subconjuntos usando solo un argumento:
m[5][1] 0.3295078
Normalmente esto no es tan útil y muchas veces difícil de leer. Sin embargo es útil notar que las matrices están acomodadas en un formato column-major por defecto. Esto significa que los elementos del vector están acomodados por columnas:
matrix(1:6, nrow=2, ncol=3) [,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6
Si quisieramos llenar una matriz por filas, usamos byrow=TRUE:
matrix(1:6, nrow=2, ncol=3, byrow=TRUE) [,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6
Podemos también hacer subconjuntos de las matrices usando los nombres de sus filas y de sus columnas en vez de usar sus índices.
Dado el siguiente código:
m <- matrix(1:18, nrow=3, ncol=6)
print(m) [,1] [,2] [,3] [,4] [,5] [,6]
[1,] 1 4 7 10 13 16
[2,] 2 5 8 11 14 17
[3,] 3 6 9 12 15 18
- ¿Cuál de los siguientes comandos extraerá los valores 11 y 14?
A. m[2,4,2,5]
B. m[2:5]
C. m[4:5,2]
D. m[2,c(4,5)]
D
Ahora introduciremos un nuevo operador para hacer subconjuntos. Existen tres funciones para hacer subconjuntos de listas. Ya las hemos visto cuando aprendimos los vectores atómicos y las matrices: [, [[ y $.
Usando [ siempre se obtiene una lista. Si quieres un subconjunto de una lista, pero no quieres extraer un elemento, entonces probablemente usarás [.
xlist <- list(a = "Software Carpentry", b = 1:10, data = head(mtcars))
xlist[1]$a
[1] "Software Carpentry"
Esto regresa una lista de un elemento.
Podemos hacer subconjuntos de elementos de la lista de la misma manera que con los vectores atómicos usando [. Las operaciones de comparación sin embargo no funcionan, ya que no son recursivas, estas probarán la condición en la estructura de datos de los elementos de la lista, y no en los elementos individuales de dichas estructuras de datos.
xlist[1:2]$a
[1] "Software Carpentry"
$b
[1] 1 2 3 4 5 6 7 8 9 10
Para extraer elementos individuales de la lista, tendrás que hacer uso de la función doble corchete: [[.
xlist[[1]][1] "Software Carpentry"
Nota que ahora el resultados es un vector, no una lista.
No puedes extraer más de un elemento al mismo tiempo:
xlist[[1:2]]Error in xlist[[1:2]]: subscript out of bounds
Tampoco puedes usarlo para saltar elementos:
xlist[[-1]]Error in xlist[[-1]]: invalid negative subscript in get1index <real>
Pero tú puedes usar los nombres para hacer subconjuntos y extraer elementos:
xlist[["a"]][1] "Software Carpentry"
La función $ es una manera abreviada para extraer elementos por nombre:
xlist$data mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
Dada la siguiente lista:
xlist <- list(a = "Software Carpentry", b = 1:10, data = head(mtcars))Usando tu conocimiento para hacer subconjuntos de listas y vectores, extrae el número 2 de xlist. Pista: el número 2 está contenido en el elemento “b” de la lista.
xlist$b[2][1] 2
xlist[[2]][2][1] 2
xlist[["b"]][2][1] 2
Dado un modelo lineal:
mod <- aov(pop ~ lifeExp, data=gapminder)Extrae los grados de libertad residuales (pista: attributes() te puede ayudar)
attributes(mod) ## `df.residual` es uno de los nombres de `mod`mod$df.residualRecordemos que las data frames son listas, por lo que aplican reglas similares. Sin embargo estos también son objetos de dos dimensiones:
[ con un argumento funcionará de la misma manera que para las listas, donde cada elemento de la lista corresponde a una columna. El objeto devuelto será una data frame:
head(gapminder[3]) pop
1 8425333
2 9240934
3 10267083
4 11537966
5 13079460
6 14880372
Similarmente, [[ extraerá una sola columna:
head(gapminder[["lifeExp"]])[1] 28.801 30.332 31.997 34.020 36.088 38.438
Con dos argumentos, [ se comporta de la misma manera que para las matrices:
gapminder[1:3,] country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
Si nuestro subconjunto es una sola fila, el resultado será una data frame (porque los elementos son de distintos tipos):
gapminder[3,] country year pop continent lifeExp gdpPercap
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
Pero para una sola columna el resultado será un vector (esto puede cambiarse con el tercer argumento, drop = FALSE).
Corrige cada uno de los siguientes errores para hacer subconjuntos de data frames:
- Extraer observaciones colectadas en el año 1957.
gapminder[gapminder$year = 1957,]- Extraer todas las columnas excepto de la 1 a la 4.
gapminder[,-1:4]- Extraer las filas donde la esperanza de vida es mayor a 80 años.
gapminder[gapminder$lifeExp > 80]- Extraer la primer fila, y la cuarta y quinta columna (
lifeExpygdpPercap).
gapminder[1, 4, 5]- Avanzado: extraer las filas que contienen información para los años 2002 y 2007
gapminder[gapminder$year == 2002 | 2007,]Corrige cada uno de los siguientes errores para hacer subconjuntos de data frames:
- Extraer observaciones colectadas en el año 1957.
# gapminder[gapminder$year = 1957,]
gapminder[gapminder$year == 1957,]- Extraer todas las columnas excepto de la 1 a la 4.
# gapminder[,-1:4]
gapminder[,-c(1:4)]- Extraer las filas donde la esperanza de vida es mayor a 80 años.
# gapminder[gapminder$lifeExp > 80]
gapminder[gapminder$lifeExp > 80,]- Extraer la primer fila, y la cuarta y quinta columna (
lifeExpygdpPercap).
# gapminder[1, 4, 5]
gapminder[1, c(4, 5)]- Avanzado: extraer las filas que contienen información para los años 2002 y 2007.
# gapminder[gapminder$year == 2002 | 2007,]
gapminder[gapminder$year == 2002 | gapminder$year == 2007,]
gapminder[gapminder$year %in% c(2002, 2007),]-
¿Por qué
gapminder[1:20]regresa un error? ¿En qué difiere degapminder[1:20, ]? -
Crea un
data.framellamadogapminder_smallque solo contenga las filas del 1 al 9 y del 19 al 23. Puedes hacerlo en uno o dos pasos.
1. gapminder es un data.frame por lo que para hacer un subconjunto necesita dos dimensiones. gapminder[1:20, ] genera un subconjunto de los datos de las primeras 20 filas y todas las columnas.
gapminder_small <- gapminder[c(1:9, 19:23),]-
Los índices en R comienzan con 1, no con 0.
-
Acceso a un elemento por posición usando
[]. -
Acceso a un rango de datos usando
[min:max]. Acceso a subconjuntos arbitrarios usando[c(...)]. -
Usar operaciones lógicas y vectores lógicos para acceder a subconjuntos de datos.
-
¿Cómo puedo hacer elecciones dependiendo de mis datos en R?
-
¿Cómo puedo repetir operaciones en R?
-
Escribir declaraciones condicionales utilizando
if()yelse(). -
Escribir y entender los bucles con
for().
Cuando estamos programando puede que queramos controlar el flujo de nuestras acciones. Esto se puede realizar estableciendo acciones que ocurran solo si se cumple una condición o un conjunto de condiciones. A su vez, podemos hacer que una acción ocurra un número determinado de veces.
Hay varias maneras de controlar el flujo en R. Para declaraciones condicionales, los enfoques más comúnmente utilizados son los constructs:
# if
if (la condición es verdad) {
realizar una acción
}
# if ... else
if (la condición es verdad) {
realizar una acción
} else { # es decir, si la condición es falsa,
realizar una acción alternativa
}Digamos, por ejemplo, que queremos que R imprima un mensaje si una variable x tiene un valor en particular:
x <- 8
if (x >= 10) {
print("x es mayor o igual que 10")
}
x[1] 8
La sentencia print “x es mayor o igual que 10” no aparece en la consola porque x no es mayor o igual a 10. Para imprimir un mensaje diferente para numeros menores a 10, podemos agregar una sentencia else
x <- 8
if (x >= 10) {
print("x es mayor o igual a 10")
} else {
print("x es menor a 10")
}[1] "x es menor a 10"
También podemos testear múltiples condiciones usando else if
x <- 8
if (x >= 10) {
print("x es mayor o igual a 10")
} else if (x > 5) {
print("x es mayor a 5, pero menor a 10")
} else{
print("x es menor a 5")
}[1] "x es mayor a 5, pero menor a 10"
Importante: cuando R evalúa las condiciones dentro de if() esta buscando elementos lógicos como TRUE o FALSE. Entender esto puede ser un dolor de cabeza para los principiantes. Por ejemplo:
x <- 4 == 3
if (x) {
"4 igual a 3"
} else {
"4 no es igual a 3"
}[1] "4 no es igual a 3"
Como podemos observar se muestra el mensaje es igual porque el vector x es FALSE
x <- 4 == 3
x[1] FALSE
Usa una declaración if() para mostrar un mensaje adecuado reportando si hay algún registro del año 2002 en el dataset gapminder. Luego haz lo mismo para el año 2012.
Primero veremos una solución al Desafío 1 que no usa la función any(). Primero obtenemos un vector lógico que describe que el elemento gapminder$year es igual a 2002:
gapminder[(gapminder$year == 2002),]Luego, contamos el número de filas del data.frame gapminder que corresponde al año 2002:
rows2002_number <- nrow(gapminder[(gapminder$year == 2002),])La presencia de cualquier registro para el año 2002 es equivalente a la petición de que rows2002_number sea mayor o igual a uno:
rows2002_number >= 1Entonces podríamos escribir:
if(nrow(gapminder[(gapminder$year == 2002),]) >= 1){
print("Se encontraron registro(s) para el año 2002.")
}Todo esto se puede hacer más rápido con any(). En dicho caso la condición lógica se puede expresar como:
if(any(gapminder$year == 2002)){
print("Se encontraron registro(s) para el año 2002.")
}¿Alguien recibió un mensaje de advertencia como este?
Error in eval(expr, envir, enclos): object 'gapminder' not found
Si tu condición se evalúa como un vector con más de un elemento lógico, la función if() todavía se ejecutará, pero solo evaluará la condición en el primer elemento. Aquí debes asegurarte que tu condición sea de longitud 1.
La función any() devolverá TRUE si hay al menos un valor TRUE dentro del vector, en caso contrario devolverá FALSE. Esto se puede usar de manera similar al operador %in%. La función all(), como el nombre sugiere, devolvera TRUE siempre y cuando todos los valores en el vector son TRUE.
Si quieres iterar sobre un conjunto de valores, el orden de la iteración es importante y debes realizar la misma operación en cada uno, un bucle for() es lo que estas buscando. Vimos los bucles for() en las lecciones anteriores de terminal. Si bien esta es la operación de bucle más flexible es también la más difícil de usar correctamente. Evita usar bucles for() a menos que el orden de la iteración sea importante como cuando el cálculo de cada iteración dependa del resultado de la iteración previa.
La estructura básica de un bucle for() es:
for(iterador in conjunto de valores){
haz alguna acción
}Por ejemplo:
for(i in 1:10){
print(i)
}[1] 1
[1] 2
[1] 3
[1] 4
[1] 5
[1] 6
[1] 7
[1] 8
[1] 9
[1] 10
El rango 1:10 crea un vector sobre la marcha; puedes iterar sobre cualquier otro vector también.
Podemos usar un bucle for() anidado con otro bucle for() para iterar sobre dos cosas a la vez.
for(i in 1:5){
for(j in c('a', 'b', 'c', 'd', 'e')){
print(paste(i,j))
}
}[1] "1 a"
[1] "1 b"
[1] "1 c"
[1] "1 d"
[1] "1 e"
[1] "2 a"
[1] "2 b"
[1] "2 c"
[1] "2 d"
[1] "2 e"
[1] "3 a"
[1] "3 b"
[1] "3 c"
[1] "3 d"
[1] "3 e"
[1] "4 a"
[1] "4 b"
[1] "4 c"
[1] "4 d"
[1] "4 e"
[1] "5 a"
[1] "5 b"
[1] "5 c"
[1] "5 d"
[1] "5 e"
En lugar de mostrar los resultados en la pantalla podríamos guardarlos en un nuevo objeto.
output_vector <- c()
for(i in 1:5){
for(j in c('a', 'b', 'c', 'd', 'e')){
temp_output <- paste(i, j)
output_vector <- c(output_vector, temp_output)
}
}
output_vector [1] "1 a" "1 b" "1 c" "1 d" "1 e" "2 a" "2 b" "2 c" "2 d" "2 e" "3 a" "3 b"
[13] "3 c" "3 d" "3 e" "4 a" "4 b" "4 c" "4 d" "4 e" "5 a" "5 b" "5 c" "5 d"
[25] "5 e"
Este enfoque puede ser útil, pero aumentar o incrementar tus resultados (es decir, construir el objeto resultante de forma incremental) es computacionalmente ineficiente por lo que conviene evitarlo cuando estés iterando a través de muchos valores.
Una de las cosas que más frecuentemente hace tropezar tanto a los principiantes como a los usuarios experimentados de R es la construcción de un objeto de resultados (vector, lista, matriz, data frame) a medida que tu bucle for progresa. Las computadoras son muy malas para manejar esto lo cual puede hacer que tus cálculos se enlentezcan rápidamente. En este caso es mejor definir un objeto de resultados vacío con las dimensiones apropiadas. Si sabes que el resultado final se almacenará en una matriz como la anterior es una buena idea crear una matriz vacía con 5 filas y 5 columnas y luego en cada iteración almacenar los resultados en la ubicación adecuada.
Una mejor manera es definir el objeto de salida (vacío) antes de completar los valores. Aunque para este ejemplo parece más complicado, es aún más eficiente.
output_matrix <- matrix(nrow=5, ncol=5)
j_vector <- c('a', 'b', 'c', 'd', 'e')
for(i in 1:5){
for(j in 1:5){
temp_j_value <- j_vector[j]
temp_output <- paste(i, temp_j_value)
output_matrix[i, j] <- temp_output
}
}
output_vector2 <- as.vector(output_matrix)
output_vector2 [1] "1 a" "2 a" "3 a" "4 a" "5 a" "1 b" "2 b" "3 b" "4 b" "5 b" "1 c" "2 c"
[13] "3 c" "4 c" "5 c" "1 d" "2 d" "3 d" "4 d" "5 d" "1 e" "2 e" "3 e" "4 e"
[25] "5 e"
Algunas veces tendrás la necesidad de repetir una operación hasta que cierta condición se cumpla. Puedes hacer esto con un bucle while().
while(mientras esta condición es verdad){
haz algo
}A modo de ejemplo aquí hay un bucle while que genera números aleatorios a partir de una distribución uniforme (la función runif() ) entre 0 y 1 hasta que obtiene uno que es menor a 0.1.
z <- 1
while(z > 0.1){
z <- runif(1)
print(z)
}Los bucles while() no siempre serán la elección apropiada. Debes ser particularmente cuidadoso de que tu condición se cumpla y no terminar en un bucle infinito.
Compara los objetos output_vector y output_vector2. ¿Son lo mismo? Si no, ¿por qué no? ¿Cómo cambiarías el último bloque de código para hacer output_vector2 igual a output_vector?
Podemos verificar si dos vectores son idénticos usando la funciónall() :
all(output_vector == output_vector2)Sin embargo todos los elementos de output_vector se pueden encontrar en output_vector2:
all(output_vector %in% output_vector2)y viceversa:
all(output_vector2 %in% output_vector)Por lo tanto, los elementos en output_vector y output_vector2 están en distinto orden. Esto es porque as.vector () genera los elementos leyendo la matriz de entrada por columnas. Si observamos output_matrix podemos notar que deseamos obtener sus elementos ordenados por filas. La solución es transponer la output_matrix. Podemos hacerlo llamando a la función de transposición t () o ingresando los elementos en el orden correcto. La primera solución requiere cambiar el original
output_vector2 <- as.vector(output_matrix)por
output_vector2 <- as.vector(t(output_matrix))La segunda solución requiere cambiar
output_matrix[i, j] <- temp_outputpor
output_matrix[j, i] <- temp_outputEscribe un script que a través de bucles recorra los datos gapminder por continente e imprima si la esperanza de vida media es menor o mayor de 50 años.
Paso 1: Queremos asegurarnos de que podamos extraer todos los valores únicos del vector continente
gapminder <- read.csv("data/gapminder-FiveYearData.csv")
unique(gapminder$continent)Paso 2: También tenemos que recorrer cada uno de estos continentes y calcular la esperanza de vida promedio para cada “subconjunto” de datos. Podemos hacer eso de la siguiente manera:
-
Recorre cada uno de los valores únicos de ‘continente’
-
Para cada valor de continente, crea una variable temporal que almacene la vida útil para ese subconjunto,
-
Regresar la expectativa de vida calculada al usuario imprimiendo el resultado:
for( iContinent in unique(gapminder$continent) ){
tmp <- mean(subset(gapminder, continent==iContinent)$lifeExp)
cat("Average Life Expectancy in", iContinent, "is", tmp, "\n")
rm(tmp)
}Paso 3: El ejercicio solo requiere que se imprima el resultado si la expectativa de vida promedio es menor a 50 o superior a 50. Por lo tanto, debemos agregar una condición ‘if’ antes de imprimir, lo cual evalúa si la expectativa de vida promedio calculada es superior o inferior a un umbral, e imprime una salida condicional en el resultado. Necesitamos corregir (3) desde arriba:
3a. Si la esperanza de vida calculada es menor que algún umbral (50 años), devuelve el continente e imprime la frase “la esperanza de vida es menor que el umbral”, de lo contrario devuelve el continente e imprime la frase “la esperanza de vida es mayor que el umbral”:
thresholdValue <- 50
for( iContinent in unique(gapminder$continent) ){
tmp <- mean(subset(gapminder, continent==iContinent)$lifeExp)
if(tmp < thresholdValue){
cat("Average Life Expectancy in", iContinent, "is less than", thresholdValue, "\n")
}
else{
cat("Average Life Expectancy in", iContinent, "is greater than", thresholdValue, "\n")
} # end if else condition
rm(tmp)
} # end for loopModifica el script del Desafío 4 para obtener resultados para cada uno de los países. En esta oportunidad que imprima si la esperanza de vida es menor que 50, se encuentra entre 50 y 70 o es mayor que 70.
Modificamos nuestra solución al Reto 3 agregando ahora dos umbrales, lowerThreshold yupperThreshold y extendiendo nuestras declaraciones if-else:
lowerThreshold <- 50
upperThreshold <- 70
for( iCountry in unique(gapminder$country) ){
tmp <- mean(subset(gapminder, country==iCountry)$lifeExp)
if(tmp < lowerThreshold){
cat("Average Life Expectancy in", iCountry, "is less than", lowerThreshold, "\n")
}
else if(tmp > lowerThreshold && tmp < upperThreshold){
cat("Average Life Expectancy in", iCountry, "is between", lowerThreshold, "and", upperThreshold, "\n")
}
else{
cat("Average Life Expectancy in", iCountry, "is greater than", upperThreshold, "\n")
}
rm(tmp)
}Error in eval(expr, envir, enclos): object 'gapminder' not found
Escribir un script que con un bucle recorra cada país en el dataset gapminder, probar si el país comienza con una ‘B’ y graficar la esperanza de vida contra el tiempo como un gráfico de líneas si la esperanza de vida media es menor de 50 años.
Lo primero que vamos a hacer es usar el comando grep que se introdujo en la lección Shell de Unix para encontrar países que comiencen con” B “.”
Si seguimos la lección de Shell de Unix podríamos vernos tentados de probar lo siguiente
grep("^B", unique(gapminder$country))Pero cuando evaluamos este comando obtenemos los índices de la variable del factor country que comienza con “B”. Para obtener los valores, debemos agregar la opción value = TRUE al comandogrep:
grep("^B", unique(gapminder$country), value=TRUE)A continuación almacenaremos estos países en una variable llamada candidateCountries y luego con un bucle recorreremos cada entrada en la variable.
Dentro del bucle se evaluará la expectativa de vida promedio para cada país y de ser menor a 50 usamos un gráfico base para trazar la evolución de la expectativa de vida promedio:
thresholdValue <- 50
candidateCountries <- grep("^B", unique(gapminder$country), value=TRUE)
for( iCountry in candidateCountries){
tmp <- mean(subset(gapminder, country==iCountry)$lifeExp)
if(tmp < thresholdValue){
cat("Average Life Expectancy in", iCountry, "is less than",
thresholdValue, "plotting life expectancy graph... \n")
with(subset(gapminder, country==iCountry),
plot(year,lifeExp,
type="o",
main = paste("Life Expectancy in", iCountry, "over time"),
ylab = "Life Expectancy",
xlab = "Year"
) # end plot
) # end with
} # end for loop
rm(tmp)
}-
Usar
ifyelsepara realizar elecciones. -
Usar
forpara operaciones repetidas.
- ¿Cómo puedo crear gráficos con calidad para publicación en R
-
Ser capaz de utilizar ggplot2 para generar gráficos con calidad para publicación
-
Entender la gramática básica de los gráficos, incluyendo estética y capas geométricas, agregando estadísticas, transformando las escalas y los colores, o dividiendo por grupos.
Graficar nuestros datos es una de las mejores maneras para explorarlos y para observar las relaciones que existen entre las variables.
En R existen tres sistemas principales encargados de hacer gráficos, el sistema de ploteo base, el paquete lattice y el paquete ggplot2.
Hoy aprenderemos acerca de ggplot2, que permite de manera sencilla crear gráficos complejos a partir de un conjunto de datos. Este paquete provee una estructura para que podamos especificar qué variables graficar, cómo deben ser presentadas y otras propiedades visuales generales (consulta la guía rápida de funciones).
De esta manera, sólo necesitamos realizar cambios mínimos si los datos sufren alguna modificación o si decidimos pasar, por ejemplo, de un gráfico de barras a un diagrama de dispersión. Esto ayuda a crear gráficos con calidad para publicación con pocos ajustes adicionales.
ggplot2 se basa en la gramática de gráficos, una idea que plantea que cualquier gráfico puede expresarse a partir de la combinación de estos componentes: un conjunto de datos, un sistema de coordenadas y un conjunto de geoms (que determinan la representación visual de los datos).
La clave para entender ggplot2 es pensar en una figura como un conjunto de capas. Esta idea podría resultarte familiar si has usado un programa de edición de imágenes como Photoshop, Illustrator o Inkscape. Esto quiere decir que los gráficos de ggplot2 se construyen paso a paso agregando nuevos elementos o capas, resultando en una gran flexibilidad para la personalización de los mismos.
Para construir un ggplot, emplearemos esta plantilla básica que puede usarse para distintos tipos de gráficos:
ggplot(data = <DATOS>, mapping = aes(<MAPEOS>)) + <GEOM_FUNCIÓN>()
- Usa la función
ggplot()para vincular el gráfico a un conjunto de datos específico a través del argumentodata. A las funciones deggplot2les gusta trabajar con datos en formato largo, es decir, una columna para cada variable y una fila para cada observación. Tener datos bien estructurados te ahorrará mucho tiempo a la hora de realizar gráficos conggplot2. Además, todos los argumentos que le pasemos a la funciónggplot()serán considerados como opciones globales en nuestro gráfico, lo cual significa que estas opciones son válidas para todas sus capas:
library("ggplot2")
ggplot(data = gapminder)- Define un mapeo (usando la función
aes()dentro deggplot()) para seleccionar las variables a graficar y especificar cómo deben ser presentadas, por ejemplo, en los ejes x/y o como característicales tales como tamaño, forma, color, etc.aes()le dice aggplotcómo se relaciona cada una de las variables en los datos con las propiedades aesthetic (estéticas) de la figura. En este caso, le decimos aggplotque queremos graficar la columna “gdpPercap” del data frame gapminder en el eje X, y la columna “lifeExp” en el eje Y. Nota que no necesitamos indicar explícitamente estas columnas en la funciónaes(e.g.x = gapminder[, "gdpPercap"]), ¡esto es debido a queggplotes suficientemente listo para buscar esa columna en los datos!:
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp))- Agrega geoms, que indican cómo se representan de los datos en el gráfico (puntos, líneas, barras).
ggplot2ofrece muchos geoms diferentes, algunos comúnmente utilizados son:
* `geom_point()` para diagramas de dispersión, gráficos de puntos, etc.
* `geom_boxplot()` para diagramas de caja y bigotes.
* `geom_line()` para líneas de tendencias, series de tiempo, etc.
Para agregar un geom, usa el operador +. ¡Podemos agregar varios geoms unidos a través de múltiples operadores +! Observa que en la figura anterior, llamar a la función ggplot no fue suficiente para obtener un gráfico, debemos agregar un geom. Podemos usar geom_point para representar visualmente la relación entre x y y como un gráfico de dispersión de puntos (scatterplot).
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()Modifica el ejemplo anterior de manera que en la figura se muestre como la esperanza de vida ha cambiado a través del tiempo:
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp)) + geom_point()Modifica el ejemplo de manera que en la figura se muestre como la esperanza de vida ha cambiado a través del tiempo:
Esta es una posible solución:
ggplot(data = gapminder, aes(x = year, y = lifeExp)) + geom_point()En los ejemplos y desafíos anteriores hemos usado la función aes para decirle al geom_point cuáles serán las posiciones x y y para cada punto. Otra propiedad aesthetic que podemos modificar es el color. Modifica el código del desafío anterior para colorear los puntos de acuerdo a la columna “continent”. ¿Qué tendencias observas en los datos? ¿Son lo que esperabas?
En los ejemplos y desafios anteriores hemos usado la función aes para decirle al geom_point cuáles serán las posiciones x y y para cada punto. Otra propiedad aesthetic que podemos modificar es el color. Modifica el código del desafío anterior para colorear los puntos de acuerdo a la columna “continent”. ¿Qué tendencias observas en los datos? ¿Son lo que esperabas?
ggplot(data = gapminder, aes(x = year, y = lifeExp, color=continent)) +
geom_point()Un gráfico de dispersión probablemente no es la mejor manera de visualizar el cambio a través del tiempo. En vez de eso, vamos a decirle a ggplot que queremos visualizar los datos como un diagrama de línea (line plot):
ggplot(data = gapminder, aes(x=year, y=lifeExp, by=country, color=continent)) +
geom_line()En vez de agregar una capa geom_point, hemos agregado una capa geom_line. Además, hemos agregado el argumento aesthetic by, el cual le dice a ggplot que debe dibujar una línea para cada país.
Pero, ¿qué pasa si queremos visualizar ambos, puntos y líneas en la misma gráfica? Simplemente tenemos que agregar otra capa al gráfico:
ggplot(data = gapminder, aes(x=year, y=lifeExp, by=country, color=continent)) +
geom_line() + geom_point()Es importante notar que cada capa se dibuja encima de la capa anterior. En este ejemplo, los puntos se han dibujado sobre las líneas. A continuación observamos una demostración:
ggplot(data = gapminder, aes(x=year, y=lifeExp, by=country)) +
geom_line(aes(color=continent)) + geom_point()En este ejemplo, el mapeo aesthetic de color se ha movido de las opciones globales de la gráfica en ggplot a la capa geom_line y, por lo tanto, ya no es válido para los puntos. Ahora podemos ver claramente que los puntos se dibujan sobre las líneas.
Hasta ahora, hemos visto cómo usar un aesthetic (como color) como un mapeo entre una variable de los datos y su representación visual. Por ejemplo, cuando usamos geom_line(aes(color=continent)), ggplot le asignará un color diferente a cada continente. Pero, ¿qué tal si queremos cambiar el color de todas las líneas a azul? Podrías pensar que geom_line(aes(color="blue")) debería funcionar, pero no es así. Dado que no queremos crear un mapeo hacia una variable específica, simplemente debemos cambiar la especificación de color afuera de la función aes(), de esta manera: geom_line(color="blue").
Intercambia el orden de las capas de los puntos y líneas del ejemplo anterior. ¿Qué sucede?
ggplot(data = gapminder, aes(x=year, y=lifeExp, by=country)) +
geom_point() + geom_line(aes(color=continent))¡Las líneas ahora están dibujadas sobre los puntos!
ggplot también facilita sobreponer modelos estadísticos a los datos. Para demostrarlo regresaremos a nuestro primer ejemplo:
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp, color=continent)) +
geom_point()En este momento es difícil ver las relaciones entre los puntos debido a algunos valores altamente atípicos de la variable GDP per capita. Podemos cambiar la escala de unidades del eje X usando las funciones de escala. Estas funciones controlan la relación entre los valores de los datos y los valores visuales de un aesthetic. También podemos modificar la transparencia de los puntos, usando la función alpha, la cual es especialmente útil cuando tienes una gran cantidad de datos fuertemente conglomerados.
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10()La función log10 aplica una transformación sobre los valores de la columna “gdpPercap” antes de presentarlos en el gráfico, de manera que cada múltiplo de 10 ahora corresponde a un incremento de 1 en la escala transformada, e.g. un GDP per capita de 1,000 se convierte en un 3 en el eje Y, un valor de 10,000 corresponde a un valor de 4 en el eje Y, y así sucesivamente. Esto facilita visualizar la dispersión de los datos sobre el eje X.
Nota que usamos geom_point(alpha = 0.5). Como menciona la sugerencia anterior, cambiar una especificación afuera de la función aes() causará que este valor sea usado para todos los puntos, que es exactamente lo que queremos en este caso. Sin embargo, como cualquier otra especificación aesthetic, alpha puede ser mapeado hacia una variable de los datos. Por ejemplo, podemos asignar una transparencia diferente a cada continente usando geom_point(aes(alpha = continent)).
Podemos ajustar una relación simple a los datos agregando otra capa, geom_smooth:
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp)) +
geom_point() + scale_x_log10() + geom_smooth(method="lm")`geom_smooth()` using formula = 'y ~ x'
Podemos hacer la línea más gruesa configurando el argumento aesthetic tamaño en la capa geom_smooth:
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp)) +
geom_point() + scale_x_log10() + geom_smooth(method="lm", size=1.5)Warning: Using `size` aesthetic for lines was deprecated in ggplot2 3.4.0.
ℹ Please use `linewidth` instead.
This warning is displayed once every 8 hours.
Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
generated.
`geom_smooth()` using formula = 'y ~ x'
Existen dos formas en las que un aesthetic puede ser especificado. Aquí configuramos el aesthetic tamaño pasándolo como un argumento a geom_smooth. Previamente en la lección habíamos usado la función aes para definir un mapeo entre alguna variable de los datos y su representación visual.
Modifica el color y el tamaño de los puntos en la capa puntos en el ejemplo anterior
Pista: No uses la función aes.
Modifica el color y el tamaño de los puntos en la capa puntos en el ejemplo anterior
Pista: No uses la función aes.
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp)) +
geom_point(size=3, color="orange") + scale_x_log10() +
geom_smooth(method="lm", size=1.5)`geom_smooth()` using formula = 'y ~ x'
Modifica tu solución al Desafío 4a de manera que ahora los puntos tengan una forma diferente y estén coloreados de acuerdo al continente incluyendo líneas de tendencia.
Pista: El argumento color puede ser usado dentro de aesthetic
Modifica tu solución al Desafío 4a de manera que ahora los puntos tengan una forma diferente y estén coloreados de acuerdo al continente incluyendo líneas de tendencia.
Pista: El argumento color puede ser usado dentro de aesthetic
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp, color = continent)) +
geom_point(size=3, shape=17) + scale_x_log10() +
geom_smooth(method="lm", size=1.5)`geom_smooth()` using formula = 'y ~ x'
Anteriormente visualizamos el cambio en la esperanza de vida a lo largo del tiempo para cada uno de los países en un solo gráfico. Como una alternativa, podemos dividir este gráfico en múltiples paneles al agregar una capa facet, enfocándonos únicamente en aquellos países con nombres que empiezan con la letra “A” o “Z”.
Empezamos por subdividir los datos. Usamos la función substr para extraer una parte de una secuencia de caracteres; extrayendo las letras que ocurran de la posición start hasta stop, inclusive, del vector gapminder$country. El operador %in% nos permite hacer múltiples comparaciones y evita que tengamos que escribir una condición muy larga para subdividir los datos (en este caso, starts.with %in% c("A", "Z") es equivalente a starts.with == "A" | starts.with == "Z")
starts.with <- substr(gapminder$country, start = 1, stop = 1)
az.countries <- gapminder[starts.with %in% c("A", "Z"), ]
ggplot(data = az.countries, aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country)La capa facet_wrap toma una “fórmula” como argumento, lo cual se indica por el símbolo ~. Esto le dice a R que debe dibujar un panel para cada valor único de la columna “country” del dataset gapminder.
Para limpiar esta figura y que quede lista para publicar necesitamos cambiar algunos elementos de texto. El eje X está demasiado saturado, y el nombre del eje Y debería ser “Esperanza de vida”, en vez del nombre que aparece para esa columna en el data frame.
Podemos hacer todo lo anterior agregando un par de capas. La capa theme controla el texto de los ejes, y el tamaño del texto. Las etiquetas de los ejes, el título del gráfico y el título para todas las leyendas pueden ser configurados utilizando la función labs. Los títulos de las leyendas son configurados haciendo referencia a los mismos nombres que utilizamos en la especificación aes. Entonces, en el siguiente ejemplo el título de la leyenda de los colores de las líneas se define utilizando color = "Continent", mientras que el título de la leyenda del color del relleno se definiría utilizando fill = "MyTitle".
ggplot(data = az.countries, aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country) +
labs(
x = "Year", # título del eje x
y = "Life expectancy", # título del eje y
title = "Figure 1", # título principal de la figura
color = "Continent" # título de la leyenda
) +
theme(axis.text.x=element_blank(), axis.ticks.x=element_blank())La función ggsave() permite exportar una gráfica creada con ggplot. Puedes especificar la dimensión y la resolución de tu gráfica ajustando los argumentos apropiados (width, height y dpi) para crear gráficas de gran calidad para publicar. Para poder guardar la gráfica de arriba, primero tenemos que asignarla a una variable lifeExp_plot, y luego decirle a ggsave que guarde la gráfica en formato png a un directorio llamado results. (Asegúrate de que tienes una carpeta llamada results/ en tu directorio de trabajo.)
lifeExp_plot <- ggplot(data = az.countries, aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country) +
labs(
x = "Year", # título del eje x
y = "Life expectancy", # título del eje y
title = "Figure 1", # título principal de la figura
color = "Continent" # título de la leyenda
) +
theme(axis.text.x=element_blank(), axis.ticks.x=element_blank())
ggsave(filename = "results/lifeExp.png", plot = lifeExp_plot, width = 12, height = 10, dpi = 300, units = "cm")Error in `ggsave()`:
! Cannot find directory 'results'.
ℹ Please supply an existing directory or use `create.dir = TRUE`.
Hay dos cosas agradables acerca de ggsave. Primero, usa la última gráfica por default, así que si omites el argumento plot, ggsave automáticamente guardará la última gráfica que creaste con ggplot. En segundo lugar, ggsave trata de determinar el formato en que quieres guardar la gráfica a partir de la extensión provista en el nombre del archivo (por ejemplo .png or .pdf). Si es necesario, puedes especificar el formato explícitamente en el argumento device.
Esta lección es una prueba de lo que puedes hacer utilizando ggplot2. RStudio proporciona una hoja de ayuda realmente útil de las diferentes capas disponibles, una documentación más extensa se encuentra disponible en el sitio web de ggplot2. Finalmente, si no tienen idea de cómo cambiar algún detalle, una búsqueda rápida en Google te llevarán a Stack Overflow donde encuentras preguntas y respuestas ¡con código reusable que puedes modificar!
Crea una gráfica de densidad de GDP per cápita, en la que el color del relleno cambie por continente.
Avanzado:
-
Transforma el eje X para visualizar mejor la dispersión de los datos.
-
Agrega una capa facet para generar gráficos de densidad con un panel para cada año.
Crea una gráfica de densidad de GDP per cápita, en la que el color del relleno cambie por continente.
Avanzado:
-
Transforma el eje X para visulaizar mejor la dispersión de los datos.
-
Agrega una capa facet para generar gráficos de densidad con un panel para cada año.
ggplot(data = gapminder, aes(x = gdpPercap, fill=continent)) +
geom_density(alpha=0.6) + facet_wrap( ~ year) + scale_x_log10()-
Usar
ggplot2para crear gráficos. -
Piensa cada gráfico como capas: estética, geometría, estadisticas, transformaciones de escala, y agrupamiento.
- ¿Cómo puedo operar sobre todos los elementos de un vector a la vez?
- Entender las operaciones vertorizadas en R.
La mayoría de las funciones en R están vectorizadas, lo que significa que la función operará sobre todos los elementos de un vector sin necesidad de hacer un bucle a través de cada elemento y actuar sobre cada uno de ellos. Esto hace la escritura de código más concisa, fácil de leer y menos propenso a errores.
x <- 1:4
x * 2[1] 2 4 6 8
La multiplicación se aplicó a cada elemento del vector.
También podemos sumar dos vectores juntos:
y <- 6:9
x + y[1] 7 9 11 13
Cada elemento de x fue sumado a su correspondiente elemento de y:
x: 1 2 3 4
+ + + +
y: 6 7 8 9
---------------
7 9 11 13Probemos esto en la columna pop del dataset gapminder.
Haz una nueva columna en el data frame gapminder que contiene la población en unidades de millones de personas. Comprueba el principio o el final del data frame para asegurar que funcionó.
Intenta esto en la columna pop del dataset gapminder.
Haz una nueva columna en el data frame gapminder que contiene población en unidades de millones de personas. Comprueba el principio o el final del data frame para asegurar que funcionó.
gapminder$pop_millions <- gapminder$pop / 1e6
head(gapminder) country year pop continent lifeExp gdpPercap pop_millions
1 Afghanistan 1952 8425333 Asia 28.801 779.4453 8.425333
2 Afghanistan 1957 9240934 Asia 30.332 820.8530 9.240934
3 Afghanistan 1962 10267083 Asia 31.997 853.1007 10.267083
4 Afghanistan 1967 11537966 Asia 34.020 836.1971 11.537966
5 Afghanistan 1972 13079460 Asia 36.088 739.9811 13.079460
6 Afghanistan 1977 14880372 Asia 38.438 786.1134 14.880372
En una sola gráfica, traza la población, en millones, en comparación con el año, para todos los países. No te preocupes en identificar qué país es cuál.
Repite el ejercicio, graficando sólo para China, India, e Indonesia. Nuevamente, no te preocupes acerca de cuál es cuál.
Recuerda tus habilidades de graficación al crear una gráfica con la población en millones en comparación con el año.
ggplot(gapminder, aes(x = year, y = pop_millions)) +
geom_point()countryset <- c("China","India","Indonesia")
ggplot(gapminder[gapminder$country %in% countryset,],
aes(x = year, y = pop_millions)) +
geom_point()Operadores de comparación, operadores lógicos y muchas otras funciones también están vectorizadas:
Operadores de Comparación
x > 2[1] FALSE FALSE TRUE TRUE
Operadores Lógicos
a <- x > 3 # o, para más claridad, a <- (x > 3)
a[1] FALSE FALSE FALSE TRUE
any() devuelve TRUE si algún elemento del vector es TRUE all() devuelve TRUE si todos los elementos del vector son TRUE
La mayoría de las funciones también operan elemento por elemento en los vectores:
Funciones
x <- 1:4
log(x)[1] 0.0000000 0.6931472 1.0986123 1.3862944
Operaciones vectorizadas en matrices:
m <- matrix(1:12, nrow=3, ncol=4)
m * -1 [,1] [,2] [,3] [,4]
[1,] -1 -4 -7 -10
[2,] -2 -5 -8 -11
[3,] -3 -6 -9 -12
Muy importante: el operador* te da una multiplicación de elemento por elemento! Para hacer multiplicación de matrices, necesitás usar el operador %*%:
m %*% matrix(1, nrow=4, ncol=1) [,1]
[1,] 22
[2,] 26
[3,] 30
matrix(1:4, nrow=1) %*% matrix(1:4, ncol=1) [,1]
[1,] 30
Para saber más sobre Álgebra de matrices, ver Quick-R reference guide
Dada la siguiente matriz:
m <- matrix(1:12, nrow=3, ncol=4)
m [,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12
Escribe lo que crees que sucederá cuando se ejecute:
-
m ^ -1 -
m * c(1, 0, -1) -
m > c(0, 20) -
m * c(1, 0, -1, 2)
¿Obtuviste la salida que esperabas? Si no, pregunta a un ayudante!
Dada la siguiente matriz:
m <- matrix(1:12, nrow=3, ncol=4)
m [,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12
Escribe lo que piensas que sucederá cuando ejecutes:
m ^ -1
m ^ -1 [,1] [,2] [,3] [,4]
[1,] 1.0000000 0.2500000 0.1428571 0.10000000
[2,] 0.5000000 0.2000000 0.1250000 0.09090909
[3,] 0.3333333 0.1666667 0.1111111 0.08333333
- m * c(1, 0, -1)
m * c(1, 0, -1) [,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 0 0 0 0
[3,] -3 -6 -9 -12
- m > c(0, 20)
m > c(0, 20) [,1] [,2] [,3] [,4]
[1,] TRUE FALSE TRUE FALSE
[2,] FALSE TRUE FALSE TRUE
[3,] TRUE FALSE TRUE FALSE
Estamos interesados en encontrar la suma de la siguiente secuencia de fracciones:
x = 1/(1^2) + 1/(2^2) + 1/(3^2) + ... + 1/(n^2)Esto sería tedioso de escribir, e imposible para valores altos de n. Usa vectorización para calcular x cuando n=100. ¿Cuál es la suma cuando n=10.000?
Estamos interesados en encontrar la suma de la siguiente secuencia de fracciones:
x = 1/(1^2) + 1/(2^2) + 1/(3^2) + ... + 1/(n^2)Esto sería tedioso de escribir, e imposible para valores altos de n. ¿Puedes usar vectorización para calcular x, cuando n=100? ¿Qué tal cuando n=10,000?
sum(1/(1:100)^2)[1] 1.634984
sum(1/(1:1e04)^2)[1] 1.644834
n <- 10000
sum(1/(1:n)^2)[1] 1.644834
Podemos obtener el mismo resultado usando una función:
inverse_sum_of_squares <- function(n) {
sum(1/(1:n)^2)
}
inverse_sum_of_squares(100)[1] 1.634984
inverse_sum_of_squares(10000)[1] 1.644834
n <- 10000
inverse_sum_of_squares(n)[1] 1.644834
- Uso de operaciones vectorizadas en lugar de bucles.
- ¿Cómo puedo escribir una nueva función en R?
-
Definir una función que usa argumentos.
-
Obtener un valor a partir de una función.
-
Revisar argumentos con
stopifnot()en las funciones. -
Probar una función.
-
Establecer valores por defecto para los argumentos de las funciones .
-
Explicar por qué debemos dividir programas en funciones pequeñas y de propósitos únicos.
Comando : Traducción
stopifnot : para si no
NULL : nulo
paste : pegar
TRUE : verdadero
FALSE : falso
source : fuente
Si tuviéramos un único conjunto de datos para analizar, probablemente sería más rápido cargar el archivo en una hoja de cálculo y usarla para graficar estadísticas simples. Sin embargo, los datos gapminder son actualizados periódicamente, y podríamos querer volver a bajar esta información actualizada más adelante y re-analizar los datos. También podríamos obtener datos similares de una fuente distinta en el futuro.
En esta lección, aprenderás cómo escribir una función de forma que seamos capaces de repetir varias operaciones con un comando único.
Las funciones reúnen una secuencia de operaciones como un todo, almacenandola para su uso continuo. Las funciones proveen:
-
un nombre que podemos recordar y usar para invocarla
-
una solución para la necesidad de recordar operaciones individuales
-
un conjunto definido de inputs y outputs esperados
-
una mayor conexión con el ambiente de programación
Como el componente básico de la mayoría de los lenguajes de programación, las funciones definidas por el usuario constituyen la “programación” de cualquier abstracción que puedas hacer. Si has escrito una función, eres ya todo un programador.
Empecemos abriendo un nuevo script de R en el directorio functions/ y nombrémosle functions-lesson.R.
my_sum <- function(a, b) {
the_sum <- a + b
return(the_sum)
}Definamos una función fahr_a_kelvin() que convierta temperaturas de Fahrenheit a Kelvin:
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}Definimos fahr_a_kelvin() asignándola al output de function. La lista de los nombres de los argumentos se encuentran entre paréntesis. Luego, el cuerpo de la función–los comandos que son ejecutados cuando se corre–se encuentran entre paréntesis curvos ({}). Los comandos en el cuerpo se indentan con dos espacios. Esto hace que el código sea legible sin afectar su funcionalidad.
Cuando utilizamos la función, los valores que definimos como argumentos se asignan a esas variables para que podamos usarlos dentro de la función. Dentro de la función, usamos un return statement para devolver un resultado a quien lo solicitó.
Una característica única de R es que el return statement no es necesario. R automáticamente devuelve cualquier variable que esté en la última línea del cuerpo de la función. Pero por claridad, nosotros explícitamente definiremos el return statement.
Tratemos de correr nuestra función. Llamamos nuestra propia función de la misma manera que llamamos cualquier otra:
# Punto de congelación del agua
fahr_to_kelvin(32)[1] 273.15
# Punto de ebullición del agua
fahr_to_kelvin(212)[1] 373.15
Escribe una función llamada kelvin_a_celsius() que toma la temperatura en grados Kelvin y devuelve la temperatura en Celsius.
Pista: Para convertir de Kelvin a Celsius se debe restar 273.15
Escribe una función llamada kelvin_a_celsius() que toma la temperatura en grados Kelvin y devuelve la temperatura en Celsius.
kelvin_to_celsius <- function(temp) {
celsius <- temp - 273.15
return(celsius)
}El poder real de las funciones proviene de mezclarlas y combinarlas en pedazos de código aún mas grandes para lograr el resultado que buscamos.
Definamos dos funciones que convertirán la temperatura de Fahrenheit a Kelvin, y de Kelvin a Celsius:
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
kelvin_to_celsius <- function(temp) {
celsius <- temp - 273.15
return(celsius)
}Define la función para convertir directamente de Fahrenheit a Celsius, reutilizando las dos funciones de arriba (o utilizando tus propias funciones si lo prefieres).
Define la función para convertir directamente de Fahrenheit a Celsius, reutilizando las dos funciones de arriba.
fahr_to_celsius <- function(temp) {
temp_k <- fahr_to_kelvin(temp)
result <- kelvin_to_celsius(temp_k)
return(result)
}Ahora que hemos empezado a apreciar cómo las funciones proporcionan una manera eficiente de hacer que el código R sea reutilizable y modular, debemos tener en cuenta que es importante garantizar que las funciones solo funcionen en los casos de uso previstos. Revisar los parámetros de las funciones está relacionado con el concepto de programación defensiva. La programación defensiva nos alienta a probar las condiciones frecuentemente y arrojar un error si algo está mal. Estas pruebas se conocen como assertion statements porque queremos asegurarnos de que una determinada condición es TRUE antes de proceder. Esto facilita la depuración porque nos dan una mejor idea de dónde se originan los errores.
Empecemos por re-examinar fahr_a_kelvin(), nuestra función para convertir temperaturas de Fahrenheit a Kelvin. Estaba definida de la siguiente manera:
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}Para que esta función trabaje como se desea, el argumento temp debe ser un valor numeric; de lo contrario, el procedimiento matemático para convertir entre las dos escalas de temperatura no funcionará. Para crear un error, podemos usar la función stop(). Por ejemplo, dado que el argumento temp debe ser un vector numeric, podríamos probarlo con un condicional if y devolver un error si la condición no se cumple. Podríamos agregar esto a nuestra función de la siguiente manera:
fahr_to_kelvin <- function(temp) {
if (!is.numeric(temp)) {
stop("temp must be a numeric vector.")
}
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}Si tuviéramos muchas condiciones o argumentos para revisar, podría llevar muchas líneas de código probarlas todas. Afortunadamente R provee la función de conveniencia stopifnot(). Podemos listar todos los requerimientos que deben ser evaluados como TRUE; stopifnot() arroja un error si encuentra uno que sea FALSE. Listar estas condiciones tiene como objetivo secundario el generar documentación extra para la función.
Hagamos la programación defensiva con stopifnot() agregando aseveraciones para probar el input a nuestra función fahr_a_kelvin().
Queremos asegurar lo siguiente: temp es un vector numérico. Lo podemos hacer de la siguiente manera:
fahr_to_kelvin <- function(temp) {
stopifnot(is.numeric(temp))
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}Aún funciona si se le da un input adecuado.
# Punto de congelación del agua
fahr_to_kelvin(temp = 32)[1] 273.15
Pero falla instantáneamente si se le da un input inapropiado.
# La métrica es un factor en lugar de numeric
fahr_to_kelvin(temp = as.factor(32))Error in fahr_to_kelvin(temp = as.factor(32)): is.numeric(temp) is not TRUE
Usar programación defensiva para asegurar que nuestra función fahr_a_celsius() arroja inmediatamente un error si el argumento temp se especifica inadecuadamente.
Extender la definición previa de nuestra función agregándole una llamada explícita a stopifnot(). Dado que fahr_a_celsius() es una composición de otras dos funciones, hacer pruebas a la función hace redundante el agregar pruebas a cada una de las dos funciones que la componen.
fahr_to_celsius <- function(temp) {
stopifnot(!is.numeric(temp))
temp_k <- fahr_to_kelvin(temp)
result <- kelvin_to_celsius(temp_k)
return(result)
}Ahora vamos a definir una función que calcula el Producto Interno Bruto (“GDP” en la base de datos, por sus siglas en inglés Gross Domestic Product) de un país a partir de los datos disponibles en nuestro conjunto de datos:
# Toma un dataset y multiplica la columna de población
# por la columna de GDP per capita
calcGDP <- function(dat) {
gdp <- dat$pop * dat$gdpPercap
return(gdp)
}Definimos calcGDP() asignándola al output de function. La lista de los nombres de los argumentos se encuentran entre paréntesis. Luego, el cuerpo de la función–las instrucciones que se ejecutan cuando se llama a la función– se encuentran entre llaves ({}).
Hemos indentado los comandos en el cuerpo con dos espacios. Esto hace que el código sea mas fácil de leer sin afectar su funcionamiento.
Cuando utilizamos la función, los valores que le pasamos se asignan como argumentos, que se convierten en variables dentro del cuerpo de la función.
Dentro de la función, usamos la función return() para obtener el resultado.
Esta función return() es opcional: R automáticamente devolverá el resultado de cualquier comando que se ejecute en la última línea de la función.
calcGDP(head(gapminder))[1] 6567086330 7585448670 8758855797 9648014150 9678553274 11697659231
Eso no es muy informativo. Agreguemos algunos argumentos más para poder extraer por año y país.
# Toma un dataset y multiplica la columna de población
# por la columna de GDP per capita
calcGDP <- function(dat, year=NULL, country=NULL) {
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
gdp <- dat$pop * dat$gdpPercap
new <- cbind(dat, gdp=gdp)
return(new)
}Si has estado escribiendo estas funciones en un script de R aparte (¡una buena idea!), puedes cargar las funciones en nuestra sesión de R usando la función source():
source("functions/functions-lesson.R")Ok, entonces están pasando muchas cosas en esta función ahora. En pocas palabras, ahora la función filtra un subconjunto de datos por año si el argumento año no está vacío, luego filtra un subconjunto de los resultados por país si el argumento país no está vacío. Luego calcula el GDP de los datos filtrados resultado de los dos pasos anteriores. La función luego agrega el valor calculado de GDP como una nueva columna en los datos filtrados y devuelve esto como el resultado final. Puedes ver que el output es mucho más informativo que un vector numérico.
Veamos qué sucede cuando especificamos el año:
head(calcGDP(gapminder, year=2007)) country year pop continent lifeExp gdpPercap gdp
12 Afghanistan 2007 31889923 Asia 43.828 974.5803 31079291949
24 Albania 2007 3600523 Europe 76.423 5937.0295 21376411360
36 Algeria 2007 33333216 Africa 72.301 6223.3675 207444851958
48 Angola 2007 12420476 Africa 42.731 4797.2313 59583895818
60 Argentina 2007 40301927 Americas 75.320 12779.3796 515033625357
72 Australia 2007 20434176 Oceania 81.235 34435.3674 703658358894
O para un país específico:
calcGDP(gapminder, country="Australia") country year pop continent lifeExp gdpPercap gdp
61 Australia 1952 8691212 Oceania 69.120 10039.60 87256254102
62 Australia 1957 9712569 Oceania 70.330 10949.65 106349227169
63 Australia 1962 10794968 Oceania 70.930 12217.23 131884573002
64 Australia 1967 11872264 Oceania 71.100 14526.12 172457986742
65 Australia 1972 13177000 Oceania 71.930 16788.63 221223770658
66 Australia 1977 14074100 Oceania 73.490 18334.20 258037329175
67 Australia 1982 15184200 Oceania 74.740 19477.01 295742804309
68 Australia 1987 16257249 Oceania 76.320 21888.89 355853119294
69 Australia 1992 17481977 Oceania 77.560 23424.77 409511234952
70 Australia 1997 18565243 Oceania 78.830 26997.94 501223252921
71 Australia 2002 19546792 Oceania 80.370 30687.75 599847158654
72 Australia 2007 20434176 Oceania 81.235 34435.37 703658358894
O ambos:
calcGDP(gapminder, year=2007, country="Australia") country year pop continent lifeExp gdpPercap gdp
72 Australia 2007 20434176 Oceania 81.235 34435.37 703658358894
Veamos paso a paso el cuerpo de la función:
calcGDP <- function(dat, year=NULL, country=NULL) {Aquí hemos agregado dos argumentos, año y país. Hemos establecido argumentos predeterminados para ambos como NULL usando el operador = en la definición de la función. Esto significa que esos argumentos tomarán esos valores a menos que el usuario especifique lo contrario.
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}Aquí verificamos si cada argumento adicional se define como null, y cuando no sea null se sobreescriben los datos almacenados en dat por un subconjunto de datos determinados por el argumento not-null.
Hacemos esto para que nuestra función sea más flexible para más adelante. Podemos pedirle que calcule el GDP para:
-
El dataset completo;
-
Un solo año;
-
Un solo país;
-
Una combinación única de año y país.
Al utilizar el operador %in%, también podemos asignarle múltiples años o países a estos argumentos.
Las funciones en R casi siempre hacen copias de los datos para operar dentro del cuerpo de una función. Cuando modificamos dat dentro de la función estamos modificando la copia del dataset gapminder almacenado en dat, y no la variable original que asignamos como el primer argumento.
Eso se llama ****pasar por valor**** y hace la escritura del código mucho más segura: puedes estar seguro que cualquier cambio que hagas dentro del cuerpo de la función, se mantendrá dentro de la función.
Otro concepto importante es el alcance: las variables (¡o funciones!) que creas o modificas dentro del cuerpo de una función sólo existen durante el tiempo de ejecución de la función. Cuando llamamos calcGDP(), las variables dat, gdp y new sólo existen dentro del cuerpo de la función. Incluso, si tenemos variables con el mismo nombre en nuestra sesión interactiva de R, éstas no son modificadas en ninguna manera cuando se ejecuta la función.
gdp <- dat$pop * dat$gdpPercap
new <- cbind(dat, gdp=gdp)
return(new)Finalmente, calculamos GDP en nuestro nuevo subconjunto de datos, y creamos una nueva dataframe con esta columna agregada. Esto significa que cuando llamamos a la función, en el resultado podemos ver el contexto de los valores GDP obtenidos, lo que es mucho mejor que nuestro primer intento cuando habíamos obtenido un vector de números.
Probar tu función GDP calculando el GDP para Nueva Zelandia (“New Zealand”) en 1987. ¿Cómo difiere del GDP de Nueva Zelandia en 1952?
calcGDP(gapminder, year = c(1952, 1987), country = "New Zealand")GDP para Nueva Zelandia en 1987: 65050008703
GDP para Nueva Zelandia en 1952: 21058193787
La función paste() puede ser usada para combinar texto, ej.:
best_practice <- c("Write", "programs", "for", "people", "not", "computers")
paste(best_practice, collapse=" ")[1] "Write programs for people not computers"
Escribir una función fence() que tome dos vectores como argumentos, llamados text y wrapper, y muestra el texto flanqueado del wrapper:
fence(text=best_practice, wrapper="***")Nota: la función paste() tiene un argumento llamado sep, que especifica el separador de texto. Por defecto es un espacio: ” “. El valor por defecto de la función paste0() es sin espacio”“.
Escribir una función fence() que toma dos vectores como argumentos, llamados text y wrapper, e imprime el texto flanqueado del wrapper:
fence <- function(text, wrapper){
text <- c(wrapper, text, wrapper)
result <- paste(text, collapse = " ")
return(result)
}
best_practice <- c("Write", "programs", "for", "people", "not", "computers")
fence(text=best_practice, wrapper="***")[1] "*** Write programs for people not computers ***"
R tiene algunos aspectos únicos que pueden ser explotados cuando se realizan operaciones más complicadas. No escribiremos nada que requiera el conocimiento de estos conceptos más avanzados. En el futuro, cuando te sientas cómodo escribiendo funciones en R, puedes aprender más leyendo el Manual de lenguaje de R o este capítulo de Advanced R Programming de Hadley Wickham.
Es importante probar las funciones así como documentarlas: la documentación ayuda, tanto a tí como a otros, a entender cuál es el propósito de la función y cómo usarla; además es importante asegurarse que la función realmente haga lo que tú piensas que hace.
Cuando recién comiences, tu flujo de trabajo probablemente luzca algo así:
-
Escribir una función
-
Comentar partes de la función para documentar su comportamiento
-
Cargar el archivo source
-
Experimentar con ella en la consola para asegurarte que se comporta tal como tu esperas.
-
Hacer los arreglos necesarios
-
Volver a probar y repetir.
La documentación formal para las funciones, escritas en archivos .Rd aparte, se transforman en la documentación que ves en los archivos de ayuda. El paquete roxygen2 le permite a los programadores de R escribir la documentación junto con el código, y luego procesarlo para generar los archivos .Rd apropiados. Quizás quieras cambiarte a este método más formal de escribir la documentación cuando empieces a escribir proyectos de R más complicados.
Pruebas automatizadas formales se pueden escribir usando el paquete testthat.
-
Usar
functionpara definir una nueva función en R. -
Usar parámetros para ingresar valores dentro de las funciones.
-
Usar
stopifnot()para revisar los argumentos de una función en R de manera flexible. -
Cargar funciones dentro de programas empleando
source().
- ¿Cómo puedo guardar gráficos y datos creados en R?
- Ser capaz de guardar gráficos y datos desde R.
Comando : Traducción
write.table : escribir tabla
Ya hemos visto como guardar el gráfico más reciente que creaste con el paquete ggplot2 usando el comando ggsave. A manera de recordatorio, aquí está el código:
ggsave("My_most_recent_plot.pdf")Puedes guardar un gráfico desde Rstudio usando el botón de ‘Export’ en la ventana de ‘Plot’. Esto te dará la opción de guardarlo como .pdf, .png, .jpg u otros formatos de imágenes.
Puede que quieras guardar los gráficos sin visualizarlos previamente en la ventana de ‘Plot’. Quizás quieras hacer un documento pdf con varias páginas: por ejemplo, cada una con un gráfico distinto. O quizás estás iterando sobre distintos subconjuntos de un archivo, graficando los datos de cada subconjunto y quieres guardar cada una de los gráficos. En estos casos obviamente no puedes detener la iteración en cada paso para dar clic en ‘Export’ para cada uno.
En dicho caso conviene usar un método más flexible. La función pdf crea un nuevo pdf del cual puedes controlar el tamaño y la resolución usando los argumentos específicos de esta función.
pdf("Life_Exp_vs_time.pdf", width=12, height=4)
ggplot(data=gapminder, aes(x=year, y=lifeExp, colour=country)) +
geom_line() +
theme(legend.position = "none")
# ¡Tienes que asegurarte de cerrar el pdf! Para ello usas el comando:
dev.off()Abre este documento y echa un vistazo.
Vuelve a escribir el comando ‘pdf’, pero esta vez emplea el término facet (sugerencia: usa facet_grid) con los mismos datos. Esto te permitirá visualizar en un gráfico los datos por continente y guardarlos en un pdf.
pdf("Life_Exp_vs_time.pdf", width = 12, height = 4)
p <- ggplot(data = gapminder, aes(x = year, y = lifeExp, colour = country)) +
geom_line() +
theme(legend.position = "none")
p
p + facet_grid(. ~continent)
dev.off()Los comandos como jpegy png entre otros son usados de manera similar para producir documentos en los correspondientes formatos.
En algún momento puede que también quieras guardar datos desde R.
Para ello podes usar la función write.table, que es muy similar a la función read.table que se presentó anteriormente.
Vamos a crear un script para limpiar datos. En este análisis, vamos a enfocarnos solamente en los datos de gapminder para Australia:
aust_subset <- gapminder[gapminder$country == "Australia",]
write.table(aust_subset,
file="cleaned-data/gapminder-aus.csv",
sep=","
)Ahora regresemos a la terminal para dar un vistazo a los datos y asegurarnos que se vean bien:
head cleaned-data/gapminder-aus.csv"country","year","pop","continent","lifeExp","gdpPercap"
"61","Australia",1952,8691212,"Oceania",69.12,10039.59564
"62","Australia",1957,9712569,"Oceania",70.33,10949.64959
"63","Australia",1962,10794968,"Oceania",70.93,12217.22686
"64","Australia",1967,11872264,"Oceania",71.1,14526.12465
"65","Australia",1972,13177000,"Oceania",71.93,16788.62948
"66","Australia",1977,14074100,"Oceania",73.49,18334.19751
"67","Australia",1982,15184200,"Oceania",74.74,19477.00928
"68","Australia",1987,16257249,"Oceania",76.32,21888.88903
"69","Australia",1992,17481977,"Oceania",77.56,23424.76683
Mmm, eso no era precisamente lo que queríamos. ¿De dónde vinieron todas esas comillas?. Los números de línea tampoco tienen sentido.
Veamos el archivo de ayuda para investigar como podemos cambiar este comportamiento.
?write.tableSalvo que configuremos otra cosa, los vectores character aparecen de forma predeterminada entre comillas cuando se guardan en un archivo. También se guardan los nombres de los renglones y las columnas.
Cambiemos esto:
write.table(
gapminder[gapminder$country == "Australia",],
file="cleaned-data/gapminder-aus.csv",
sep=",", quote=FALSE, row.names=FALSE
)Ahora echemos de nuevo un vistazo a los datos usando nuestras habilidades en la terminal:
head cleaned-data/gapminder-aus.csvcountry,year,pop,continent,lifeExp,gdpPercap
Australia,1952,8691212,Oceania,69.12,10039.59564
Australia,1957,9712569,Oceania,70.33,10949.64959
Australia,1962,10794968,Oceania,70.93,12217.22686
Australia,1967,11872264,Oceania,71.1,14526.12465
Australia,1972,13177000,Oceania,71.93,16788.62948
Australia,1977,14074100,Oceania,73.49,18334.19751
Australia,1982,15184200,Oceania,74.74,19477.00928
Australia,1987,16257249,Oceania,76.32,21888.88903
Australia,1992,17481977,Oceania,77.56,23424.76683
¡Ahora se ve mejor!
Escribe un script para limpiar estos datos, filtrando los datos de gapminder que fueron colectados desde 1990.
Usa este script para guardar este nuevo subconjunto de datos en el directorio cleaned-data.
write.table(
gapminder[gapminder$year > 1990, ],
file = "cleaned-data/gapminder-after1990.csv",
sep = ",", quote = FALSE, row.names = FALSE
)-
Guardar gráficos desde RStudio usando el botón de ‘Export’.
-
Usar
write.tablepara guardar datos tabulares.
- ¿Cómo puedo hacer diferentes cálculos sobre diferentes conjuntos de dato?
- Estar apto a usar la estrategia divide-aplica-combina para el análisis de datos.
Previamente vimos como puedes usar funciones para simplificar tu código. Definimos la función calcGDP, la cual toma el dataset gapminder, y multiplica la columna de población por la columna GDP per cápita. También definimos argumentos adicionales de modo que pudiéramos filtrar por "year" o por "country":
# Toma un dataset y multiplica la columna population con
# la columna GDP per cápita.
calcGDP <- function(dat, year=NULL, country=NULL) {
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
gdp <- dat$pop * dat$gdpPercap
new <- cbind(dat, gdp=gdp)
return(new)
}Una tarea común que encontrarás mientras trabajes con datos, es que querrás hacer cálculos en diferentes grupos sobre los datos. En el ejemplo de arriba, simplemente calculamos el GDP por multiplicar dos columnas juntas. ¿Pero qué tal si queremos calcular la media GDP por continente?
Podríamos ejecutar calcGDP y entonces tomar la media de cada continente:
withGDP <- calcGDP(gapminder)
mean(withGDP[withGDP$continent == "Africa", "gdp"])[1] 20904782844
mean(withGDP[withGDP$continent == "Americas", "gdp"])[1] 379262350210
mean(withGDP[withGDP$continent == "Asia", "gdp"])[1] 227233738153
Pero esto no es muy bonito. Sí, por usar una función, has reducido substancialmente la cantidad de repeticiones. Esto es bonito. Pero aún hay repeticiones. La repetición te costará tiempo, tanto ahora como más tarde, y potencialmente introducirás algunos errores desagradables.
Podriamos escribir una nueva función que sea flexible como calcGDP, pero esta también requiere una gran cantidad de esfuerzo y pruebas para hacerlo bien.
El problema abstracto que estamos encontrando aquí es conocido como “divide-aplica-combina (split-apply-combine)”:
Nosotros queremos dividir (split) nuestros datos dentro de grupos, en este caso continentes, aplicar (apply) algunos cálculos sobre este grupo y, opcionalmente, combinar (combine) los resultados más tarde.
Para aquellos que han usado antes R, es posible que estén familiarizados con la familia de funciones apply. Mientras que las funciones integradas de R funcionan, vamos a introducirte a otro método para resolver el problema “split-apply-combine”. El paquete plyr proporciona un conjunto de funciones que encontramos más amigables de usar para resolver este problema.
Instalamos este paquete en un desafío anterior. Vamos a cargarlo ahora:
library("plyr")Plyr tiene una función para operar sobre listas o **lists** , **data.frames** y **arrays** (matrices, o vectores n-dimensional). Cada función realiza:
-
Una operación de división (splitting).
-
Aplica (apply) una función sobre cada una de las partes a la vez.
-
Recombina (recombine) los datos de salida como un simple objeto de datos.
Las funciones se nombran en función de la estructura de datos que esperan como entrada, y la estructura de datos que desea devolver como salida: [a]rray, [l]ist, o [d]ata.frame. La primera letra corresponde a la estructura de datos de entrada, la segunda letra a la estructura de datos de salida, y luego el resto de la función se llama “ply”.
Esto nos da 9 funciones básicas **ply. Hay adicionalmente un árbol de funciones las cuales solo realizarán la división y aplicación de los pasos y ningún paso combinado. Ellas son nombradas por sus datos de entrada y representan una salida nula con un _ (Ver tabla)
Note que el uso de “array” de plyr es diferente a R, un array en ply puede incluir a vector o matriz.
Cada una de las funciones de xxply (daply,ddply, llply,laply, …) tiene la misma estructura y 4 características clave:
xxply(.data, .variables, .fun)-
La primera letra del nombre de la función da el tipo de la entrada y el segundo da el tipo de la salida.
-
.data– Es el objeto de datos a ser procesado -
.variables– identifica la variable para hacer la división -
.fun– Da la función a ser llamada para cada piez
Ahora podemos calcular rápidamente la media GDP por "continent":
ddply(
.data = calcGDP(gapminder),
.variables = "continent",
.fun = function(x) mean(x$gdp)
) continent V1
1 Africa 20904782844
2 Americas 379262350210
3 Asia 227233738153
4 Europe 269442085301
5 Oceania 188187105354
Veamos el código anterior:
-
La función
ddplyrecibe de entrada undata.frame(La función empieza con una d) y regresa otrodata.frame(la 2da letra es una d) -
El primer argumento que dimos fue el
data.frameen el que queríamos operar: en este caso, los datos delgapminder. Primero llamamos a la funcióncalcGDPpara agregar la columnagdpa nuestra variable data. -
El segundo argumento indica nuestro criterio para dividir: En este caso la columna
"continent". Ten en cuenta que le dimos el nombre de la columna, no los valores de la columna como habíamos hecho previamente con los subconjuntos.Plyrse encarga de los detalles de la implementación por ti. -
El tercer argumento es la función que queremos aplicar a cada grupo de datos. Tenemos que definir nuestra propia función corta aquí: cada subconjunto de datos va almacenado en
x, el primer argumento de la función. Esta es una función anónima: no la hemos definido en otra parte y no tiene nombre. Solo existe en el ámbito de nuestro llamado addply.
¿Qué pasa si queremos un tipo diferente de estructura de datos de salida?:
dlply(
.data = calcGDP(gapminder),
.variables = "continent",
.fun = function(x) mean(x$gdp)
)$Africa
[1] 20904782844
$Americas
[1] 379262350210
$Asia
[1] 227233738153
$Europe
[1] 269442085301
$Oceania
[1] 188187105354
attr(,"split_type")
[1] "data.frame"
attr(,"split_labels")
continent
1 Africa
2 Americas
3 Asia
4 Europe
5 Oceania
Llamamos a la misma función otra vez, pero cambiamos la segunda letra por una l, asi que la salida fue regresada como una lista.
Podemos especificar múltiples columnas para agrupar:
ddply(
.data = calcGDP(gapminder),
.variables = c("continent", "year"),
.fun = function(x) mean(x$gdp)
) continent year V1
1 Africa 1952 5992294608
2 Africa 1957 7359188796
3 Africa 1962 8784876958
4 Africa 1967 11443994101
5 Africa 1972 15072241974
6 Africa 1977 18694898732
7 Africa 1982 22040401045
8 Africa 1987 24107264108
9 Africa 1992 26256977719
10 Africa 1997 30023173824
11 Africa 2002 35303511424
12 Africa 2007 45778570846
13 Americas 1952 117738997171
14 Americas 1957 140817061264
15 Americas 1962 169153069442
16 Americas 1967 217867530844
17 Americas 1972 268159178814
18 Americas 1977 324085389022
19 Americas 1982 363314008350
20 Americas 1987 439447790357
21 Americas 1992 489899820623
22 Americas 1997 582693307146
23 Americas 2002 661248623419
24 Americas 2007 776723426068
25 Asia 1952 34095762661
26 Asia 1957 47267432088
27 Asia 1962 60136869012
28 Asia 1967 84648519224
29 Asia 1972 124385747313
30 Asia 1977 159802590186
31 Asia 1982 194429049919
32 Asia 1987 241784763369
33 Asia 1992 307100497486
34 Asia 1997 387597655323
35 Asia 2002 458042336179
36 Asia 2007 627513635079
37 Europe 1952 84971341466
38 Europe 1957 109989505140
39 Europe 1962 138984693095
40 Europe 1967 173366641137
41 Europe 1972 218691462733
42 Europe 1977 255367522034
43 Europe 1982 279484077072
44 Europe 1987 316507473546
45 Europe 1992 342703247405
46 Europe 1997 383606933833
47 Europe 2002 436448815097
48 Europe 2007 493183311052
49 Oceania 1952 54157223944
50 Oceania 1957 66826828013
51 Oceania 1962 82336453245
52 Oceania 1967 105958863585
53 Oceania 1972 134112109227
54 Oceania 1977 154707711162
55 Oceania 1982 176177151380
56 Oceania 1987 209451563998
57 Oceania 1992 236319179826
58 Oceania 1997 289304255183
59 Oceania 2002 345236880176
60 Oceania 2007 403657044512
daply(
.data = calcGDP(gapminder),
.variables = c("continent", "year"),
.fun = function(x) mean(x$gdp)
) year
continent 1952 1957 1962 1967 1972
Africa 5992294608 7359188796 8784876958 11443994101 15072241974
Americas 117738997171 140817061264 169153069442 217867530844 268159178814
Asia 34095762661 47267432088 60136869012 84648519224 124385747313
Europe 84971341466 109989505140 138984693095 173366641137 218691462733
Oceania 54157223944 66826828013 82336453245 105958863585 134112109227
year
continent 1977 1982 1987 1992 1997
Africa 18694898732 22040401045 24107264108 26256977719 30023173824
Americas 324085389022 363314008350 439447790357 489899820623 582693307146
Asia 159802590186 194429049919 241784763369 307100497486 387597655323
Europe 255367522034 279484077072 316507473546 342703247405 383606933833
Oceania 154707711162 176177151380 209451563998 236319179826 289304255183
year
continent 2002 2007
Africa 35303511424 45778570846
Americas 661248623419 776723426068
Asia 458042336179 627513635079
Europe 436448815097 493183311052
Oceania 345236880176 403657044512
Puedes usar estas funciones en lugar de ciclos for (y generalmente es mas rápido). Para remplazar un ciclo ‘for’, pon el código que estaba en el cuerpo del ciclo for dentro de la función anónima.
d_ply(
.data=gapminder,
.variables = "continent",
.fun = function(x) {
meanGDPperCap <- mean(x$gdpPercap)
print(paste(
"La media GDP per cápita para", unique(x$continent),
"es", format(meanGDPperCap, big.mark=",")
))
}
)[1] "La media GDP per cápita para Africa es 2,193.755"
[1] "La media GDP per cápita para Americas es 7,136.11"
[1] "La media GDP per cápita para Asia es 7,902.15"
[1] "La media GDP per cápita para Europe es 14,469.48"
[1] "La media GDP per cápita para Oceania es 18,621.61"
La función format puede ser usada para imprimir los valores numéricos
“bonitos” en los mensajes.
Calcula el promedio de vida esperado por "continent". ¿Quién tiene el promedio mas alto? ¿Quién tiene el mas pequeño?
Calcula el promedio de vida esperado por "continent" y "year". ¿Quién tiene el promedio mas grande y mas corto en 2007? ¿Quién tiene el cambio mas grande entre 1952 y 2007?
Calcula la diferencia en la media de vida esperada entre los años 1952 y 2007 a partir de la salida del desafío 2 usando una de las funciones plyr.
Sin ejecutarlos, cuál de los siguientes calculará el promedio de la esperanza de vida por continente:
ddply(
.data = gapminder,
.variables = gapminder$continent,
.fun = function(dataGroup) {
mean(dataGroup$lifeExp)
}
)ddply(
.data = gapminder,
.variables = "continent",
.fun = mean(dataGroup$lifeExp)
)ddply(
.data = gapminder,
.variables = "continent",
.fun = function(dataGroup) {
mean(dataGroup$lifeExp)
}
)adply(
.data = gapminder,
.variables = "continent",
.fun = function(dataGroup) {
mean(dataGroup$lifeExp)
}
)- Uso del paquete
plyrpara dividir datos, aplicar funciones sobre subconjuntos, y combinar los resultados
- ¿Cómo manipular data frames sin repetir lo mismo una y otra vez?
-
Ser capaces de usar las seis principales acciones de manipulación de data frames con pipes en
dplyr. -
Comprender cómo combinar
group_by()ysummarize()para obtener resúmenes de datasets. -
Ser capaces de analizar un subconjunto de datos usando un filtrado lógico.
Comando : Traducción
filter : filtrar
select : seleccionar
group_by : agrupar
summarize : resumir
count : contar
mean : media
mutate : mutar
La manipulación de data frames significa distintas cosas para distintos investigadores. A veces queremos seleccionar ciertas observaciones (filas) o variables (columnas), otras veces deseamos agrupar los datos en función de una o más variables, o queremos calcular valores estadísticos de un conjunto. Podemos hacer todo ello usando las habituales operaciones básicas de R:
mean(gapminder[gapminder$continent == "Africa", "gdpPercap"])[1] 2193.755
mean(gapminder[gapminder$continent == "Americas", "gdpPercap"])[1] 7136.11
mean(gapminder[gapminder$continent == "Asia", "gdpPercap"])[1] 7902.15
Pero esto no es muy elegante porque hay demasiada repetición. Repetir cosas cuesta tiempo, tanto en el momento de hacerlo como en el futuro, y aumenta la probabilidad de que se produzcan desagradables bugs (errores).
Afortunadamente, el paquete dplyr proporciona un conjunto de funciones (consulta la guía rápida ) extremadamente útiles para manipular data frames y así reducir el número de repeticiones , la probabilidad de cometer errores y el número de caracteres que hay que escribir. Como valor extra, puedes encontrar que la gramática de dplyr es más fácil de entender.
Aquí vamos a revisar 6 de sus funciones más usadas, así como a usar los pipes (%>%) para combinarlas.
-
select() -
filter() -
group_by() -
summarize() -
mutate()
Si no has instalado antes este paquete, hazlo del siguiente modo:
install.packages('dplyr')Ahora vamos a cargar el paquete:
library("dplyr")Si por ejemplo queremos continuar el trabajo con sólo unas pocas de las variables de nuestro data frame podemos usar la función select(). Esto guardará sólo las variables que seleccionemos.
year_country_gdp <- select(gapminder,year,country,gdpPercap)Si ahora investigamos year_country_gdp veremos que sólo contiene el año, el país y la renta per cápita. Arriba hemos usado la gramática ‘normal’, pero la fortaleza de dplyr consiste en combinar funciones usando pipes. Como la gramática de las pipes es distinta a todo lo que hemos visto antes en R, repitamos lo que hemos hecho arriba, pero esta vez usando pipes.
year_country_gdp <- gapminder %>% select(year,country,gdpPercap)Para ayudarte a entender por qué lo hemos escrito así, vamos a revisarlo por partes. Primero hemos llamado al data frame “gapminder” y se lo hemos pasado al siguiente paso, que es la función select(), usando el símbolo del pipe %>%. En este caso no especificamos qué objeto de datos vamos a usar en la función select() porque esto se obtiene del resultado de la instrucción previa a el pipe. Dato curioso: es muy posible que te hayas encontrado con pipes antes en la terminal de unix. En R el símbolo del pipe es %>%, mientras que en la terminal es |, pero el concepto es el mismo.
Si ahora queremos continuar con lo de arriba, pero sólo con los países europeos, podemos combinar select y filter.
year_country_gdp_euro <- gapminder %>%
filter(continent=="Europe") %>%
select(year,country,gdpPercap)Escribe un único comando (que puede ocupar varias líneas e incluir pipes) que produzca un data frame y que tenga los valores africanos correspondientes a lifeExp, country y year, pero no de los otros continentes. ¿Cuántas filas tiene dicho data frame y por qué?
year_country_lifeExp_Africa <- gapminder %>%
filter(continent=="Africa") %>%
select(year,country,lifeExp)Al igual que la vez anterior, primero le pasamos el data frame “gapminder” a la función filter() y luego le pasamos la versión filtrada del data frame a la función select(). Nota: El orden de las operaciones es muy importante en este caso. Si usamos primero select(), la función filter() no habría podido encontrar la variable “continent” porque la habríamos eliminado en el paso previo.
Se suponía que teníamos que reducir las repeticiones causantes de errores de lo que se puede hacer con el R básico, pero hasta ahora no lo hemos conseguido porque tendríamos que repetir lo escrito arriba para cada continente. En lugar de filter(), que solamente deja pasar las observaciones que se ajustan a tu criterio (continent = Europe en lo escrito arriba), podemos usar group_by(), que esencialmente usará cada uno de los criterios únicos que podrías haber usado con filter().
str(gapminder)'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...
str(gapminder %>% group_by(continent))gropd_df [1,704 × 6] (S3: grouped_df/tbl_df/tbl/data.frame)
$ country : chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int [1:1704] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num [1:1704] 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr [1:1704] "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num [1:1704] 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num [1:1704] 779 821 853 836 740 ...
- attr(*, "groups")= tibble [5 × 2] (S3: tbl_df/tbl/data.frame)
..$ continent: chr [1:5] "Africa" "Americas" "Asia" "Europe" ...
..$ .rows : list<int> [1:5]
.. ..$ : int [1:624] 25 26 27 28 29 30 31 32 33 34 ...
.. ..$ : int [1:300] 49 50 51 52 53 54 55 56 57 58 ...
.. ..$ : int [1:396] 1 2 3 4 5 6 7 8 9 10 ...
.. ..$ : int [1:360] 13 14 15 16 17 18 19 20 21 22 ...
.. ..$ : int [1:24] 61 62 63 64 65 66 67 68 69 70 ...
.. ..@ ptype: int(0)
..- attr(*, ".drop")= logi TRUE
Se puede observar que la estructura del data frame obtenido por group_by() (grouped_df) no es la misma que la del data frame original gapminder(data.fram). Se puede pensar en un grouped_df como en una list donde cada item in la list es un data.frame que contiene únicamente las filas que corresponden a un valor particular de continent (en el ejemplo mostrado).
Lo visto arriba no es muy sofisticado, pero group_by() es más interesante y útil si se usa en conjunto con summarize(). Esto nos permitirá crear nuevas variables usando funciones que se aplican a cada uno de los data frames específicos para cada continente. Es decir, usando la función group_by() dividimos nuestro data frame original en varias partes, a las que luego podemos aplicarles funciones (por ejemplo, mean() o sd()) independientemente con summarize().
gdp_bycontinents <- gapminder %>%
group_by(continent) %>%
summarize(mean_gdpPercap=mean(gdpPercap))continent mean_gdpPercap
<fctr> <dbl>
1 Africa 2193.755
2 Americas 7136.110
3 Asia 7902.150
4 Europe 14469.476
5 Oceania 18621.609Esto nos ha permitido calcular la renta per cápita media para cada continente, pero puede ser todavía mucho mejor.
Calcula la esperanza de vida media por país. ¿Qué país tiene la esperanza de vida media mayor y cuál la menor?
lifeExp_bycountry <- gapminder %>%
group_by(country) %>%
summarize(mean_lifeExp=mean(lifeExp))
lifeExp_bycountry %>%
filter(mean_lifeExp == min(mean_lifeExp) | mean_lifeExp == max(mean_lifeExp))# A tibble: 2 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5
2 Sierra Leone 36.8
Otro modo de hacer esto es usando la función arrange() del paquete dplyr, que distribuye las filas de un data frame en función del orden de una o más variables del data frame. Tiene una sintaxis similar a otras funciones del paquete dplyr. Se puede usar desc() dentro de arrange() para ordenar de modo descendente.
lifeExp_bycountry %>%
arrange(mean_lifeExp) %>%
head(1)# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Sierra Leone 36.8
lifeExp_bycountry %>%
arrange(desc(mean_lifeExp)) %>%
head(1)# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5
La función group_by() nos permite agrupar en función de varias variables. Vamos a agrupar por year y continent.
gdp_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap))`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
Esto ya es bastante potente, pero puede ser incluso mejor. Puedes definir más de una variable en summarize().
gdp_pop_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap),
sd_gdpPercap=sd(gdpPercap),
mean_pop=mean(pop),
sd_pop=sd(pop))`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
Una operación muy habitual es contar el número de observaciones que hay en cada grupo. El paquete dplyr tiene dos funciones relacionadas muy útiles para ello.
Por ejemplo, si queremos comprobar el número de países que hay en el conjunto de datos para el año 2002 podemos usar la función count(). Dicha función toma el nombre de una o más columnas que contienen los grupos en los que estamos interesados y puede opcionalmente ordenar los resultados en modo descendente si añadimos sort = TRUE.
gapminder %>%
filter(year == 2002) %>%
count(continent, sort = TRUE) continent n
1 Africa 52
2 Asia 33
3 Europe 30
4 Americas 25
5 Oceania 2
Si necesitamos usar en nuestros cálculos el número de observaciones obtenidas, la función n() es muy útil. Por ejemplo, si queremos obtener el error estándar de la esperanza de vida por continente:
gapminder %>%
group_by(continent) %>%
summarize(se_pop = sd(lifeExp)/sqrt(n()))# A tibble: 5 × 2
continent se_pop
<chr> <dbl>
1 Africa 0.366
2 Americas 0.540
3 Asia 0.596
4 Europe 0.286
5 Oceania 0.775
También se pueden encadenar juntas varias operaciones de resumen, como en el caso siguiente, en el que calculamos el minimum, maximum, mean y se de la esperanza de vida por país para cada continente:
gapminder %>%
group_by(continent) %>%
summarize(
mean_le = mean(lifeExp),
min_le = min(lifeExp),
max_le = max(lifeExp),
se_le = sd(lifeExp)/sqrt(n()))# A tibble: 5 × 5
continent mean_le min_le max_le se_le
<chr> <dbl> <dbl> <dbl> <dbl>
1 Africa 48.9 23.6 76.4 0.366
2 Americas 64.7 37.6 80.7 0.540
3 Asia 60.1 28.8 82.6 0.596
4 Europe 71.9 43.6 81.8 0.286
5 Oceania 74.3 69.1 81.2 0.775
También se pueden crear nuevas variables antes (o incluso después) de resumir la información usando mutate().
gdp_pop_bycontinents_byyear <- gapminder %>%
mutate(gdp_billion=gdpPercap*pop/10^9) %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap),
sd_gdpPercap=sd(gdpPercap),
mean_pop=mean(pop),
sd_pop=sd(pop),
mean_gdp_billion=mean(gdp_billion),
sd_gdp_billion=sd(gdp_billion))`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
La creación de nuevas variables se puede conectar con una condición lógica. Una simple combinación de mutate y ifelse facilita el filtrado solo allí donde se necesita: en el momento de crear algo nuevo. Esta combinación es fácil de leer y es un modo rápido y potente de descartar ciertos datos (incluso sin cambiar la dimensión conjunta del data frame) o para actualizar valores dependiendo de la condición utilizada.
## manteniendo todos los datos pero "filtrando" según una determinada condición
# calcular renta per cápita sólo para gente con una esperanza de vida por encima de 25
gdp_pop_bycontinents_byyear_above25 <- gapminder %>%
mutate(gdp_billion = ifelse(lifeExp > 25, gdpPercap * pop / 10^9, NA)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
## actualizando sólo si se cumple una determinada condición
# para esperanzas de vida por encima de 40 años, el GDP que se espera en el futuro es escalado
gdp_future_bycontinents_byyear_high_lifeExp <- gapminder %>%
mutate(gdp_futureExpectation = ifelse(lifeExp > 40, gdpPercap * 1.5, gdpPercap)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
mean_gdpPercap_expected = mean(gdp_futureExpectation))`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
En la función de creación de gráficas vimos cómo hacer una figura con múltiples paneles añadiendo una capa de paneles separados (facet panels). Aquí está el código que usamos (con algunos comentarios extra):
# Obtener la primera letra de cada país
starts.with <- substr(gapminder$country, start = 1, stop = 1)
# Filtrar países que empiezan con "A" o "Z"
az.countries <- gapminder[starts.with %in% c("A", "Z"), ]
# Construir el gráfico
ggplot(data = az.countries, aes(x = year, y = lifeExp, color = continent)) +
geom_line() + facet_wrap( ~ country)Error in ggplot(data = az.countries, aes(x = year, y = lifeExp, color = continent)): could not find function "ggplot"
Este código construye la gráfica correcta, pero también crea algunas variables starts.with y az.countries) que podemos no querer usar para nada más. Del mismo modo que usamos %>% para pasar datos con pipes a lo largo de una cadena de funciones dplyr, podemos usarlo para pasarle datos a ggplot(). Como %>% sustituye al primer argumento de una función, no necesitamos especificar el argumento data= de la función ggplot(). Combinando funciones de los paquetes dplyr y ggplot podemos hacer la misma figura sin crear ninguna nueva variable y sin modificar los datos.
gapminder %>%
# Get the start letter of each country
mutate(startsWith = substr(country, start = 1, stop = 1)) %>%
# Filter countries that start with "A" or "Z"
filter(startsWith %in% c("A", "Z")) %>%
# Make the plot
ggplot(aes(x = year, y = lifeExp, color = continent)) +
geom_line() +
facet_wrap( ~ country)Error in ggplot(., aes(x = year, y = lifeExp, color = continent)): could not find function "ggplot"
Las funciones del paquete dplyr también nos ayudan a simplificar las cosas, por ejemplo, combinando los primeros dos pasos:
gapminder %>%
# Filter countries that start with "A" or "Z"
filter(substr(country, start = 1, stop = 1) %in% c("A", "Z")) %>%
# Make the plot
ggplot(aes(x = year, y = lifeExp, color = continent)) +
geom_line() +
facet_wrap( ~ country)Error in ggplot(., aes(x = year, y = lifeExp, color = continent)): could not find function "ggplot"
Calcula la esperanza de vida media en 2002 de dos países seleccionados al azar para cada continente. Luego distribuye los nombres de los continentes en orden inverso. Pista: Usa las funciones arrange() y sample_n() del paquete dplyr, tienen una sintaxis similar a las demás funciones del paquete dplyr.
lifeExp_2countries_bycontinents <- gapminder %>%
filter(year==2002) %>%
group_by(continent) %>%
sample_n(2) %>%
summarize(mean_lifeExp=mean(lifeExp)) %>%
arrange(desc(mean_lifeExp))-
Usar el paquete
dplyrpara manipular data frames. -
Usar
select()para seleccionar variables de un data frame. -
Usar
filter()para seleccionar datos basándose en los valores. -
Usar
group_by()ysummarize()para trabajar con subconjuntos de datos. -
Usar
mutate()para crear nuevas variables.
- ¿Cómo puedo cambiar el formato de los data frames?
- Entender los conceptos de formatos de datos largo y ancho y poder convertirlos al otro formato usando
tidyr.
Las investigadoras a menudo quieren manipular sus datos del formato “ancho” al “largo”, o viceversa. El formato “largo” es donde:
-
cada columna es una variable
-
cada fila es una observación
En el formato “largo”, generalmente tienes una columna para la variable observada y las otras columnas son variables de ID.
Para el formato “ancho”, cada fila es un tema, por ejemplo un lugar o un paciente. Tendrás múltiples variables de observación, que contienen el mismo tipo de datos, para cada tema. Estas observaciones pueden ser repetidas a lo largo del tiempo, o puede ser la observación de múltiples variables (o una mezcla de ambos). Para algunas aplicaciones, es preferible el formato “ancho”. Sin embargo, muchas de las funciones de R han sido diseñadas para datos de formato “largo”. Este tutorial te ayudará a transformar tus datos de manera eficiente, independientemente del formato original.
Estos formatos de datos afectan principalmente a la legibilidad. Para los humanos, el formato “ancho” es a menudo más intuitivo ya que podemos ver más de los datos en la pantalla debido a su forma. Sin embargo, el formato “largo” es más legible para las máquinas y está más cerca al formateo de las bases de datos. Las variables de ID en nuestros marcos de datos son similares a los campos en una base de datos y las variables observadas son como los valores de la base de datos.
Primero instala los paquetes necesarios, tidyr y dplyr. Si aún no lo has hecho, puedes también instalar el grupo de paquetes tidyverse que contiene varios paquetes incluyendo tidyr y dplyr.
#install.packages("tidyr")
#install.packages("dplyr")Ahora carga los paquetes usando library.
library("tidyr")
library("dplyr")Primero, veamos la estructura structure del data frame gapminder:
str(gapminder)'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...
¿Crees que el data frame gapminder tiene un formato largo, ancho o algo intermedio?
El data frame gapminder tiene un formato intermedio. No es completamente largo porque tiene múltiples observaciones por cada variable (pop,lifeExp,gdpPercap).
A veces tenemos múltiples tipos de observaciones, como con el data frame gapminder. Entonces tendremos formatos de datos mixtos entre “largo” y “ancho”. Nosotros tenemos 3 “variables de identificación” (continente,país, año) y 3 “variables de observación” (pop, lifeExp,gdpPercap). Generalmente, es preferible que los datos estén en este formato intermedio en la mayoría de los casos, a pesar de no tener TODAS las observaciones en una sola columna. Esto es por que las 3 variables de observación tienen unidades diferentes (y cada una corresponde a una columna con su propio tipo de datos).
A menudo queremos hacer operaciones matemáticas con valores que usen las mismas unidades, esto facilita el uso de funciones en R, que a menudo se basan en vectores. Por ejemplo, si utilizamos el formato largo y calculamos un promedio de todos los los valores de población pop, esperanza de vida lifeExp y el PIB gdpPercap este resultado no tendría sentido, ya que devolvería una incongruencia de valores con 3 unidades incompatibles. La solución es que primero manipulamos los datos agrupando (ver la lección sobre dplyr), o cambiamos la estructura del marco de datos. Nota: Algunas funciones de gráficos en R (por ejemplo con ggplot2) realmente funcionan mejor con los datos de formato ancho.
Hasta ahora, hemos estado utilizando el conjunto de datos gapminder original muy bien formateado, pero los datos “reales” (es decir, nuestros propios datos de investigación) nunca estarán tan bien organizados. Veamos un ejemplo con la versión de formato ancho del conjunto de datos gapminder.
Descarga la versión ancha de los datos de gapminder desde [aquí] (https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/gh-pages/_episodes_rmd/data/gapminder_wide.csv) y guarda el archivo csv en tu carpeta de datos.
Cargaremos el archivo de datos para verlo. Nota: no queremos que las columnas de caracteres sean convertidas a factores, por lo que usamos el argumento stringsAsFactors = FALSE para deshabilitar eso, más información en la ayuda ?read.csv ().
gap_wide <- read.csv("data/gapminder_wide.csv", stringsAsFactors = FALSE)
str)'data.frame': 142 obs. of 38 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : int 31287142 10866106 7026113 1630347 12251209 7021078 15929988 4048013 8835739 614382 ...
$ pop_2007 : int 33333216 12420476 8078314 1639131 14326203 8390505 17696293 4369038 10238807 710960 ...
El primer paso es formatear los datos de ancho a largo. Usando el paquete tidyr y la función gather() podemos juntar las variables de observación en una sola variable.
gap_long <- gap_wide %>%
gather(obstype_year, obs_values, starts_with("pop"),
starts_with("lifeExp"), starts_with("gdpPercap"))
str(gap_long)'data.frame': 5112 obs. of 4 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ obstype_year: chr "pop_1952" "pop_1952" "pop_1952" "pop_1952" ...
$ obs_values : num 9279525 4232095 1738315 442308 4469979 ...
Aquí hemos utilizado la sintaxis con pipes (%>%) igual a como lo que estábamos usando en el lección anterior con dplyr. De hecho, estos son compatibles y puedes usar una mezcla de las funciones tidyr y dplyr.
Dentro de gather(), primero nombramos la nueva columna para la nueva variable de ID (obstype_year), el nombre de la nueva variable de observación conjunta (obs_value), luego los nombres de la antigua variable de observación. Nosotros podríamos tener escritas todas las variables de observación, pero como lo hacíamos en la función select() (ver la lección de dplyr), podemos usar el argumento starts_with() para seleccionar todas las variables que comiencen con la cadena de caracteres deseada. Reunir o gather también permite la sintaxis alternativa del uso del símbolo - para identificar qué variables queremos excluir (por ejemplo, las variables de identificación o ID)
gap_long <- gap_wide %>% gather(obstype_year, obs_values, -continent, -country)
str(gap_long)'data.frame': 5112 obs. of 4 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ obstype_year: chr "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" ...
$ obs_values : num 2449 3521 1063 851 543 ...
Eso puede parecer trivial con este data frame en particular, pero a veces tienes una variable de identificación ID y 40 variables de observación, con varios nombres de variables irregulares. La flexibilidad que nos da tidyr ¡es un gran ahorro de tiempo!
Ahora, obstype_year en realidad contiene información en dos partes, la observación tipo (pop,lifeExp, o gdpPercap) y el año year. Podemos usar la función separate() para dividir las cadenas de caracteres en múltiples variables.
gap_long <- gap_long %>% separate(obstype_year, into = c("obs_type", "year"), sep = "_")
gap_long$year <- as.integer(gap_long$year)Usando el data frame gap_long, calcula el promedio de esperanza de vida, población, y PIB por cada continente. Ayuda: usa group_by() y summarize() que son las funciones de dplyr que aprendiste en el episodio anterior.
gap_long %>%
group_by(continent, obs_type) %>%
summarize(means = mean(obs_values))`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
# A tibble: 15 × 3
# Groups: continent [5]
continent obs_type means
<chr> <chr> <dbl>
1 Africa gdpPercap 2194.
2 Africa lifeExp 48.9
3 Africa pop 9916003.
4 Americas gdpPercap 7136.
5 Americas lifeExp 64.7
6 Americas pop 24504795.
7 Asia gdpPercap 7902.
8 Asia lifeExp 60.1
9 Asia pop 77038722.
10 Europe gdpPercap 14469.
11 Europe lifeExp 71.9
12 Europe pop 17169765.
13 Oceania gdpPercap 18622.
14 Oceania lifeExp 74.3
15 Oceania pop 8874672.
Siempre es bueno detenerse y verificar el trabajo. Entonces, usemos el opuesto de gather() para separar nuestras variables de observación con la función spread(). Para expandir nuestro objeto gap_long() al formato intermedio original o al formato ancho usaremos esta nueva función. Comencemos con el formato intermedio.
gap_normal <- gap_long %>% spread(obs_type,obs_values)
dim(gap_normal)[1] 1704 6
dim(gapminder)[1] 1704 6
names(gap_normal)[1] "continent" "country" "year" "gdpPercap" "lifeExp" "pop"
names(gapminder)[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"
Ahora tenemos un marco de datos intermedio gap_normal con las mismas dimensiones que el gapminder original, pero el orden de las variables es diferente. Arreglemos eso antes de comprobar si son iguales con la función all.equal().
gap_normal <- gap_normal[,names(gapminder)]
all.equal(gap_normal,gapminder)[1] "Component \"country\": 1704 string mismatches"
[2] "Component \"pop\": Mean relative difference: 1.634504"
[3] "Component \"continent\": 1212 string mismatches"
[4] "Component \"lifeExp\": Mean relative difference: 0.203822"
[5] "Component \"gdpPercap\": Mean relative difference: 1.162302"
head(gap_normal) country year pop continent lifeExp gdpPercap
1 Algeria 1952 9279525 Africa 43.077 2449.008
2 Algeria 1957 10270856 Africa 45.685 3013.976
3 Algeria 1962 11000948 Africa 48.303 2550.817
4 Algeria 1967 12760499 Africa 51.407 3246.992
5 Algeria 1972 14760787 Africa 54.518 4182.664
6 Algeria 1977 17152804 Africa 58.014 4910.417
head(gapminder) country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134
Ya casi, el data frame original está ordenado por country, continent, y year. Entonces probemos con la función arrange().
gap_normal <- gap_normal %>% arrange(country, continent, year)
all.equal(gap_normal, gapminder)[1] TRUE
¡Muy bien! Pasamos del formato largo al intermedio y no hemos tenido ningún error en nuestro código.
Ahora pasemos a convertir del formato largo al ancho. En el formato ancho, nosotros mantendremos el país y el continente como variables de ID y esto va expandir las observaciones en las tres métricas (pop,lifeExp, gdpPercap) y año (year). Primero nosotros necesitamos crear etiquetas apropiadas para todas nuestras nuevas variables (combinaciones de métricas*año) y también necesitamos unificar nuestras variables de ID para simplificar el proceso de definir el nuevo objeto gap_wide.
gap_temp <- gap_long %>% unite(var_ID, continent, country, sep = "_")
str(gap_temp)'data.frame': 5112 obs. of 4 variables:
$ var_ID : chr "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ obs_type : chr "gdpPercap" "gdpPercap" "gdpPercap" "gdpPercap" ...
$ year : int 1952 1952 1952 1952 1952 1952 1952 1952 1952 1952 ...
$ obs_values: num 2449 3521 1063 851 543 ...
gap_temp <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_")
str(gap_temp)'data.frame': 5112 obs. of 3 variables:
$ ID_var : chr "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ var_names : chr "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" ...
$ obs_values: num 2449 3521 1063 851 543 ...
Usando la función unite() tenemos ahora un único ID que es la combinación de continent, country, y así definimos nuestras nuevas variables. Ahora podemos usar ese resultado con la función spread().
gap_wide_new <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year,sep = "_") %>%
spread(var_names, obs_values)
str(gap_wide_new)'data.frame': 142 obs. of 37 variables:
$ ID_var : chr "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num 33333216 12420476 8078314 1639131 14326203 ...
Crea un formato de datos gap_super_wide mediante la distribución por países, año y las tres métricas. Ayuda este nuevo data frame sólo debe tener cinco filas.
gap_super_wide <- gap_long %>%
unite(var_names, obs_type, year, country, sep = "_") %>%
spread(var_names, obs_values)Ahora tenemos un gran data frame con formato ‘ancho’, pero el ID_var podría ser más mejor, hay que separarlos en dos variables con separate()
gap_wide_betterID <- separate(gap_wide_new, ID_var, c("continent", "country"), sep = "_")
gap_wide_betterID <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_") %>%
spread(var_names, obs_values) %>%
separate(ID_var, c("continent", "country"), sep = "_")
str(gap_wide_betterID)'data.frame': 142 obs. of 38 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num 33333216 12420476 8078314 1639131 14326203 ...
¡Muy bien hiciste el cambio de formato de ida y vuelta!
-
[R para Ciencia de datos] (r4ds.had.co.nz)
-
[Hoja de trucos para la conversión de datos] (https://www.rstudio.com/wp-content/uploads/2015/02/data-wrangling-cheatsheet.pdf)
-
[Introducción a tidyr] (https://cran.r-project.org/web/packages/tidyr/vignettes/tidy-data.html)
-
[Manipulación de datos con R y RStudio] (https://www.rstudio.com/resources/webinars/data-wrangling-with-r-and-rstudio/)
-
Usar el paquete
tidyrpara cambiar el diseño de los data frames. -
Usar
gather()para invertir del formato ancho al formato largo. -
Usar
spread()para invertir del formato largo al formato ancho.
- ¿Cómo puedo integrar programas e informes?
-
Valor de informes reproducibles
-
Conceptos básicos de Markdown
-
Fragmentos de código en R
-
Opciones de fragmentos
-
Código R en línea
-
Otros formatos de salida
Los analistas de datos tienden a escribir muchos informes, describiendo sus análisis y resultados, para sus colaboradores o para documentar su trabajo para referencia futura.
Es común que muchos nuevos usuarios comiencen escribiendo una rutina R con todos sus trabajos. Luego simplemente envien el análisis por un correo electrónico a su colaborador, describiendo los resultados y adjuntando el script y varios gráficos. Éste método puede ser problemático, ya que al discutir los resultados, a menudo hay confusión sobre qué gráfico corresponde a cuál resultado.
Escribir informes formales, con Word o LaTeX, puede simplificar esto, al incorporar el análisis y los resultados a un documento único. Pero arreglando el formato del documento para hacer que las figuras se vean bien y corregir saltos de página rebeldes puede ser tedioso y llevar mucho tiempo.
Crear una página web (como un archivo html) usando R markdown hace que el todo sea mas sencillo. El reporte puede ser muy largo, así que figuras altas que no cabrían normalmente en una página, pueden incluirse en tamaño original, ya que el lector las puede ver simplemente desplazando la página. Dar formato es simple y fácil de modificar, por lo que podemos dedicar más tiempo a nuestros análisis y no a la redacción de informes.
Idealmente, dichos informes de análisis son documentos reproducibles: Si se descubre un error, o si se agregan algunos temas adicionales a los datos, puedes volver a compilar el informe y obtener los resultados, nuevo o corregido (en lugar de tener que reconstruir figuras, pegarlas en un documento de Word y luego editar manualmente varios resultados detallados).
La herramienta clave para R es knitr, que te permite crear un documento que es una mezcla de texto y algunos fragmentos de código. Cuando el documento es procesado por knitr, los fragmentos del código R serán ejecutados, y los gráficos u otros resultados serán insertados.
Este tipo de idea ha sido llamado “programación literaria”.
knitr te permite mezclar básicamente cualquier tipo de texto con cualquier tipo de código, pero te recomendamos que uses R Markdown, que mezcla Markdown con R. Markdown es un ligero lenguaje de marcado para crear páginas web y también otros formatos.
Dentro de R Studio, haz clic en Archivo; Nuevo archivo; R Markdown y obtendrás un cuadro de diálogo parecido a éste:
Puedes mantener el valor predeterminado (salida HTML), pero dale un título.
El fragmento inicial de texto contiene instrucciones para R: le das un título, autor y fecha, y díle que va a querer producir una salida html (en otras palabras, una página web).
---
title: "Documento inicial **R Markdown**"
author: "Cristian Farias"
date: "09 de Octubre de 2024"
output: html_document
---
Puedes eliminar cualquiera de esos campos si no los quieres incluidos. Las comillas dobles no son estrictamente necesarias en este caso. Pero en su mayoría son necesarias si deseas incluir dos puntos en el título.
RStudio crea el documento con un texto de ejemplo para ayudarte a empezar. Observa a continuación que hay fragmentos como
summary(cars)
Estos son fragmentos de código R que serán ejecutados por knitr y reemplazados por sus resultados. Más sobre esto más tarde.
También fíjate en la dirección web que se coloca entre corchetes angulares (<>) así como los asteriscos dobles en **Knit**. Esto es Markdown.
Markdown es un sistema para escribir páginas web marcando el texto tanto como lo haría en un correo electrónico en lugar de escribir código html. El texto marcado es convertido a html, reemplazando las marcas con el código HTML.
Por ahora, borremos todas las cosas que están ahí y escribamos un poco de markdown.
Haces las cosas en negrita usando dos asteriscos, como esto: ** negrita **, y haces cosas italicas usando guiones bajos, como esto: _italics_.
Puedes hacer una lista con viñetas escribiendo una lista con guiones o asteriscos, como esta:
* negrita con doble asterisco
* itálica con guiones bajos
* tipografía estilo código fuente con acento inverso/grave
o así:
- negrita con doble asterisco
- itálica con guiones bajos
- tipografía estilo código fuente con acento inverso/grave
Cada uno aparecerá como:
-
negrita con doble asterisco
-
itálica con guiones bajos
-
tipografía estilo código fuente con acento inverso/grave
Puedes usar el método que prefieras (guiones o asteriscos), pero se consistente. Esto mantiene la legibilidad del código.
Puedes hacer una lista numerada simplemente usando números. Puedes incluso usar el mismo número una y otra vez si lo deseas:
1. negrita con doble asterisco
1. itálica con guiones bajos
1. tipografía estilo código fuente con acento inverso/grave
Esto aparecerá como:
-
negrita con doble asterisco
-
itálica con guiones bajo
-
tipografía estilo código fuente con acento inverso/grave
Puedes crear encabezados de sección de diferentes tamaños iniciando una línea con un cierto número de símbolos #:
# Título
## Sección principal
### Subsección
#### Sub-subsección
Tú compilas el documento R Markdown a una página web html haciendo clic en el “Knit HTML” en la esquina superior izquierda. Y ten en cuenta el pequeño signo de interrogación junto a él; haz clic en el signo de interrogación y obtendrá un “Markdown Quick Reference” (con la sintaxis Markdown) así como para la documentación de RStudio en R Markdown.
Crea un nuevo documento R Markdown. Elimina todos los fragmentos de código R y escribe un poco de Markdown (algunas secciones, algunos textos en itálica, y una lista de ítemes).
Convierte el documento a una página web.
Puedes hacer un hipervínculo como éste: [text to show](http://the-web-page.com).
Puedes incluir un archivo de imagen como éste: 
Puedes hacer subíndices (por ejemplo, F2) con F~2 y superíndices (p. F^2^) con F^2^.
Si sabes cómo escribir ecuaciones en [LaTeX] (http://www.latex-project.org/), te alegrará saber que puedes usar $ $ y $$ $$ para insertar ecuaciones matemáticas, como $E = mc^2$ y
$$y = \mu + \sum_{i=1}^p \beta_i x_i + \epsilon$$
Markdown es interesante y útil, pero el poder real proviene de la mezcla entre markdown y fragmentos de código R. Esto es R Markdown. Cuando procesado, el código R se ejecutará; si producen figuras, las figuras se insertarán en el documento final.
Los fragmentos del código principal se ven así:
{r load_data} gapminder Es decir, coloca un fragmento de código R entre{r chunk_name} y ```. Es una buena idea darle a cada fragmento un nombre, ya que te ayudarán a corregir los errores y, si alugnos gráficos son producidos, los nombres de archivo estarán basados en el nombre del fragmento de código que los produjo.
Agrega fragmentos de código para
-
Cargar el paquete ggplot2
-
Leer los datos del gapminder
-
Crear un gráfico
Cuando presionas el botón “Knit HTML”, el documento R Markdown es procesado por knitr y un documento Markdown simple es producido (así como, potencialmente, un conjunto de archivos de figuras): el código R es ejecutado y reemplazado por ambas la entrada y la salida; si las figuras son producidas, se incluyen enlaces a esas figuras.
Los documentos Markdown y figura son entones procesados por la herramienta pandoc, que convierte el archivo Markdown en un archivo html, con las figuras embebidas.
Hay una variedad de opciones quen afectan la forma en que los fragmentos de código son tratado
-
Usa
echo=FALSEpara evitar que se muestre el código en sí. -
Usa
results="hide"para evitar que se impriman los resultados. -
Usa
eval=FALSEpara tener el código mostrado pero no evaluado. -
Usa
warning=FALSEymessage=FALSEpara ocultar cualquier advertencia o mensajes producidos. -
Usa
fig.heightyfig.widthpara controlar el tamaño de las figuras producidas (en pulgadas).
Entonces podrías escribir:
```{r load_libraries, echo=FALSE, message=FALSE} library("dplyr")
library("ggplot2")
```
A menudo habrá opciones particulares que querrás usar repetidamente; para esto, puede establecer las opciones de fragnento global, de esta forma:
knitr::opts_chunk$set(fig.path="Figs/", message=FALSE, warning=FALSE,echo=FALSE, results="hide", fig.width=11)
La opción fig.path define dónde se guardarán las figuras. El / aquí es realmente importante; sin él, las figuras se guardarían en el lugar estándar, pero solo con los nombres que están con Figs.
Si tienes varios archivos R Markdown en un directorio común, es posible que quieras usar fig.path para definir prefijos separados para los nombres de archivo de figura , como fig.path="Figs/cleaning-" y fig.path="Figs/analysis-".
También puedes convertir R Markdown en un documento PDF o Word. Haz clic en el pequeño triángulo junto al botón “Knit HTML” para obtener un menú desplegable. O podrías poner pdf_document o word_document en el encabezado del archivo.
La creación de documentos .pdf puede requerir la instalación de algún programa adicional. Si eso es requerido, se detalla en un mensaje de error.
Tex para Windows está disponible aquí.
Tex for mac está disponible aquí.
-
Informes mixtos escritos en R Markdown usando un programa escrito en R.
-
Especificar opciones de fragmento para controlar el formateo.
-
Usar
knitrpara convertir estos documentos en PDF y en otros formatos
- ¿Cómo puedo escribir software que otras personas puedan usar?
- Describir las mejores prácticas para escribir software en R y explicar la justificación de cada una.
La parte más importante de escribir código es hacer que sea legible y comprensible. Quieres que otra persona pueda tener tu código y comprenda lo que hace: la mayoría de las veces, esta persona serás tú 6 meses después y si no es comprensible, te estarás culpando por no haber hecho las cosas bien.
Cuando empiezas a programar, tus comentarios a menudo describen lo que hace un comando, ya que aún te encuentras en aprendizaje y pueden ayudarte a aclarar conceptos y recordártelos más tarde. Sin embargo, estos comentarios no son particularmente útiles, sobre todo cuando no recuerdes qué problema estabas tratando de resolver con tu código. Intenta incluir comentarios que te digan por qué estás resolviendo un problema y qué problema es. El cómo puede venir después de eso: es un detalle de implementación del que idealmente no deberías preocuparte.
Nuestra recomendación es que debes separar tus funciones de tus scripts y almacenarlos en un archivo separado ‘source’ que llamarás cuando abras la sesión R en tu proyecto. Este enfoque es bueno porque te deja un script ordenado y un repositorio de funciones útiles que se pueden cargar en cualquier script en tu proyecto. También te permite agrupar funciones relacionadas fácilmente.
Al principio la resolución de problemas y la escritura de funciones pueden ser tareas que te resulten desalentadoras debido a la falta de experiencia en programación. Trata de dividir tu problema en bloques digeribles y preocúpate por los detalles de la implementación más adelante. Es recomendable seguir dividiendo el problema en funciones cortas de un solo propósito.
¡Asegúrate de probar tus funciones!
Las funciones permiten una fácil reutilización dentro de en proyecto. Si identificas bloques de código similares en tu proyecto son en general candidatos a ser convertidos en funciones.
Si tus cálculos se realizan a través de una serie de funciones, entonces el proyecto se vuelve más modular y más fácil de cambiar. Este es, especialmente el caso para el cual una entrada particular siempre da una salida particular.
Manten un mismo estilo en todo tu código.
-
Documenta qué y por qué, no cómo.
-
Divide los programas en funciones cortas de un solo propósito.
-
Escribe pruebas re-ejecutables.
-
No repitas tu código.
-
Se coherente en la nomenclatura, indentación y otros aspectos del estilo.
El objetivo de esta lección es enseñar a las personas programadoras principiantes a escribir códigos modulares y adoptar buenas prácticas en el uso de R para el análisis de datos. R nos provee un conjunto de paquetes desarrollados por terceros que se usan comúnmente en diversas disciplinas científicas para el análisis estadístico. Encontramos que muchos científicos que asisten a los talleres de Software Carpentry utilizan R y quieren aprender más. Nuestros materiales son relevantes ya que proporcionan a los asistentes una base sólida en los fundamentos de R y enseñan las mejores prácticas del cómputo científico: desglose del análisis en módulos, automatización tareas y encapsulamiento.
Ten en cuenta que este taller se enfoca en los fundamentos del lenguaje de programación R y no en el análisis estadístico.
A lo largo de este taller se utilizan una variedad de paquetes desarrolados por terceros, los cuales no son necesariamente los mejores ni se encuentran explicadas todas sus funcionalidades, pero son paquetes que consideramos útiles y han sido elegidos principalmente por su facilidad de uso.
Entender que los archivos estan organizados en directorios (folders). Entender que las computadoras guardan datos e instrucciones (programas, scripts) en archivos. Saber como acceder a los archivos que no están en el directorio de trabajo, especificando el path.
Esta lección supone que tienes el software R y RStudio instalado en tu computadora.
R se puede descargar aquí.
RStudio es un entorno de desarrollo integrado para R. Se puede descargar aquí. Necesitarás la versión de escritorio para tu computadora.